En Perú, el carcinoma hepatocelular (CHC) ocupa el tercer lugar en incidencia entre los tumores del sistema digestivo, y tiene una alta tasa de mortalidad, 17.7 por 100,000 habitantes. La mayoría de los casos reportados no presentan la epidemiología clínica clásica del CHC observado en otras partes del mundo. Además, se ha identificado que estos pacientes presentan un perfil transcriptómico distinto y un proceso tumoral singular, sugiriendo un proceso particular de hepatocarcinogénesis en una fracción de la población peruana.
ObjetivoEl presente estudio busca comprender la implicancia clínica y biológica del perfil epigenético (metilación) y de expresión de los genes (transcriptómico) del CHC en los pacientes peruanos.
MétodosSe evaluó el perfil de transcriptómico y de metilación de ADN de hígado y CHC en 74 pacientes peruanos.
ResultadosEl agrupamiento por edades mostró una mayor metilación del ADN en los pacientes jóvenes con CHC, en contraste no se observaron diferencias en el perfil transcriptómico. Adicionalmente, también se evidenció una alta prevalencia del virus de hepatitis B (VHB) (> 90%) en los pacientes jóvenes con CHC. El análisis de enriquecimiento en ambos perfiles moleculares demostró que PRC2 es posiblemente uno de los principales actores moleculares en este proceso tumoral hepático en pacientes peruanos.
ConclusiónEl CHC peruano presenta un perfil molecular único, asociado a la presencia del VHB, y con una hipermetilación global del ADN asociado a células hepáticas indiferenciadas o a una reprogramación celular.
Hepatocellular carcinoma (HCC) is the third most frequent cancer of digestive tract tumors in Peru, with a high mortality rate of 17.7 per 100,000 inhabitants. A significant number of HCC cases in Peru do not follow the classic clinical epidemiology of the disease described in other parts of the world. Those patients present with a distinct transcriptome profile and a singular tumor process, suggesting a particular type of hepatocarcinogenesis in a portion of the Peruvian population.
AimOur aim was to understand the clinical and biologic involvement of the epigenetic profile (methylation) and gene expression (transcriptome) of HCC in Peruvian patients.
MethodsHCC and liver transcriptome and DNA methylation profiles were evaluated in 74 Peruvian patients.
ResultsWhen grouped by age, there was greater DNA methylation in younger patients with HCC but no differences with respect to the transcriptomic profile. A high prevalence of the hepatitis B virus (HBV) (> 90%) was also observed in the younger patients with HCC. Enrichment analyses in both molecular profiles pinpointed PRC2 as an important molecular effector of that liver tumor process in Peruvian patients.
ConclusionHCC in Peruvian patients has a unique molecular profile, associated with the presence of HBV, as well as overall DNA hypermethylation related to undifferentiated liver cells or cellular reprogramming.
En la actualidad, el cáncer de hígado es considerado un problema de salud pública en todo el mundo debido a su alta mortalidad, mal pronóstico, y frecuentes recaídas1. Es el sexto tipo de cáncer con mayor incidencia y el cuarto en mortalidad con más de 800,000 casos y 780,000 fallecidos a nivel mundial. Adicionalmente, entre 75 y 85% de los casos de cáncer de hígado primario son carcinoma hepatocelular (CHC)2.
El CHC es considerado un cáncer con un cuadro agresivo, siendo su principal factor de riesgo la cirrosis hepática (80-90%), ocasionada por altos consumos de alcohol, infecciones crónicas con los virus de hepatitis B y C (VHB y VHC), u otras condiciones metabólicas como la esteatohepatitis no alcohólica3,4. Sin embargo, también existen factores ambientales de riesgo, como la exposición a micotoxinas (aflatoxina B1)5, pesticidas6 y metaloides (arsénico)7. Los múltiples factores que originan el CHC resultan en una gran diversidad de patrones moleculares asociados al nivel del ADN y de la transcripción genética. En el 2015, Zucman-Rossi et al. propusieron la primera clasificación molecular consenso, con dos grupos principales de CHC: i) proliferativo y ii) no-proliferativo8. Estos grupos se caracterizan por diferentes mecanismos de carcinogénesis hepática (por ejemplo, factores de riesgo, firmas de expresión genética y vías de activación específicas). Adicionalmente, es importante mencionar que, a nivel epigenético, el CHC presenta generalmente una pérdida global de la metilación (hipometilación) del ADN, similar a lo observado en otros carcinomas9,10.
En América del Sur, la alta incidencia del CHC observada en el Perú lo ubica en el segundo lugar para este tipo de cáncer, superado solo por Bolivia11. Sin embargo, una fracción muy significativa de pacientes peruanos con CHC no han desarrollado previamente una cirrosis12–15. En los últimos 10 años se ha demostrado a diferentes niveles (epidemiológico, clínico, patológico, genético, transcriptómico, epigenético, etc.) que la población peruana con CHC presenta características particulares no reportadas previamente con esta magnitud en otras regiones del mundo. Las características clínicas más relevantes de los pacientes con CHC atendidos al Instituto Nacional de Enfermedades Neoplásicas (INEN) son: i) una bimodalidad con respecto a la edad con una mediana de 44 años (dos grupos etarios: ≤ 44 y>44); ii) la gran mayoría de pacientes presentan tumores gigantescos (> 10 cm); iii) aproximadamente 90% de pacientes no presentan cuadros asociados a una cirrosis; y iv) los niveles de alfa-fetoproteína (AFP) sérica son extremadamente elevados12,16.
Desde el punto de vista genético-molecular, el CHC peruano presenta un perfil de mutaciones del ADN, representado principalmente por inserciones y deleciones (indels), cuando los espectros de mutaciones del CHC descritos en la literatura con pacientes de otras partes del mundo corresponden mayormente a mutaciones transicionales y transversionales13. La caracterización del perfil transcriptómico del CHC peruano muestra una identidad molecular mixta, con características proliferativas y no-proliferativas a la vez15. Finalmente, estudios epigenéticos de este cáncer han demostrado un aumento global de la metilación (hipermetilación) del ADN, contrastando una vez más con lo reportado en otras regiones (hipometilación)9,10,15.
No obstante, ciertas características clínicas y genético-moleculares del CHC en la población peruana no han sido exploradas hasta el momento, por lo que el presente estudio explora las implicancias demográficas, clínicas y biológicas de los perfiles transcriptómico y epigenético del CHC peruano, utilizando un enfoque de análisis diferente a lo reportado anteriormente.
Material y métodosDiseño y población de estudioLos datos de los perfiles transcriptómico (n = 39) y metilación (n = 70), así como las características clínico-patológicas de pacientes peruanos diagnosticados con CHC y programados para resección hepática entre los años 2006 y 2016 en el INEN fueron obtenidos a partir de un estudio previo y descargados desde la base de datos Gene Expression Omnibus (GEO)15. Brevemente, ninguna de las muestras colectadas proviene de pacientes que hayan recibido quimioterapia antes de la resección hepática, en acuerdo a las recomendaciones de la Asociación Europea para el Estudio del Hígado (EASL)17. Las muestras fueron colectadas en pares, un tejido de CHC y un tejido de hígado adyacente no-tumoral (HNT) extraído del margen quirúrgico (≥ 2 cm). Aproximadamente 50 mg de cada tipo de tejido (CHC y HNT) fueron flash congelados y almacenados a -150°C en el Banco de Tejidos Tumorales del INEN antes del análisis. Las variables consideradas en el presente estudio incluyen datos demográficos, de laboratorio, histológicos y de la infección por VHB. Los detalles de cada variable se muestran en la tabla S1.
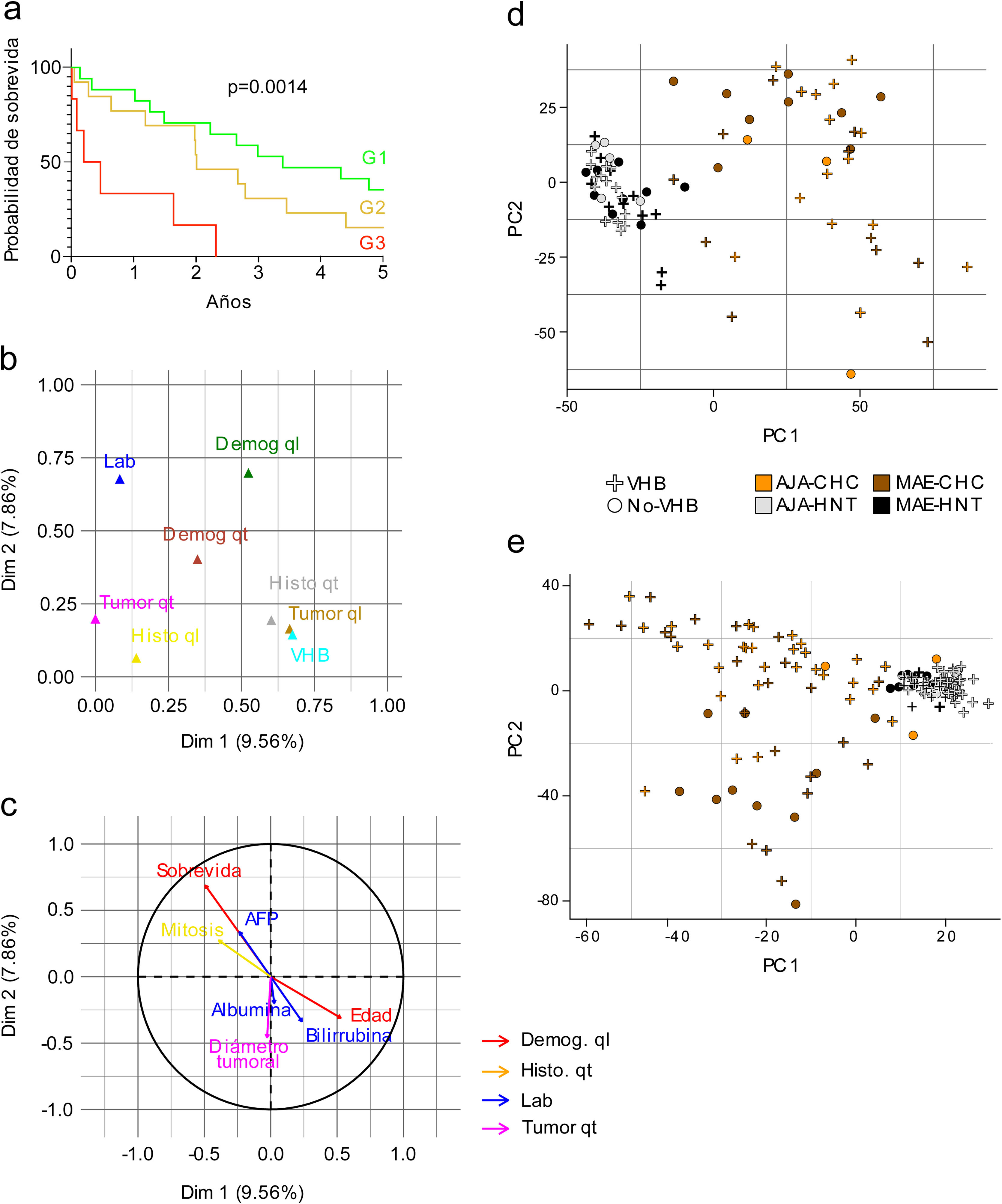
Nomograma preoperativo de predicción de sobrevidaLos pacientes fueron clasificados de acuerdo con el nomograma de pronóstico de supervivencia para tumores > 10 cm18. Las variables preoperativas consideradas en este nomograma son: i) presencia de cirrosis; ii) múltiples lesiones tumorales; iii) invasión vascular macroscópica y iv) ruptura espontánea de tumor. El puntaje final permite clasificar las muestras en tres grupos de sobrevida tal que: G1 con una alta probabilidad de supervivencia a largo plazo (0 puntos), G2 con 60% de probabilidad de recurrencia dentro de los 12 meses (entre uno y 100 puntos) y G3 con la más alta mortalidad postoperatoria (> 100 puntos).
Detección del ADN de virus de hepatitis BLa detección del ADN de VHB en los tejidos fue realizada como se describió previamente19. Brevemente, el ADN genómico se extrajo mediante el método de fenol-cloroformo y la cuantificación se realizó utilizando el kit dsDNA en la plataforma Qubit® (Invitrogen, Waltham, MA, EE. UU.). Todas las muestras se examinaron para detectar la presencia de ADN del VHB utilizando el sistema de PCR QX200™ Droplet Digital™ (Bio-Rad, Hercules, CA, EE. UU.). La secuenciación de VHB se realizó utilizando el kit de secuenciación BigDye™ Terminator v3.1 (Applied Biosystems, Waltham, MA, EE. UU.). La filogenia del VHB se determinó utilizando el modelo de dos parámetros de Kimura y el método de neighbor joining en la versión 7 del software Molecular Evolutionary Genetics Analysis (MEGA, University Park, PENN, EE. UU.). Los datos de las secuencias se pueden descargar desde el European Bioinformatics Institute (EBI) con código de acceso PRJEB21100 (https://www.ebi.ac.uk/ena/data/view/PRJEB21100).
Análisis del transcriptomaEl perfil transcriptómico se analizó a partir de un grupo de muestras de CHC peruanos obtenidos de la base de datos Gene Expression Omnibus (GEO, Bethesda, MD, EE. UU.), bajo los códigos GSE136247 y GSE11158015. El transcriptoma de estas muestras se obtuvo utilizando los microarreglos GeneChip™ Human Transcriptome Array 2.0 y el GeneChip™ WT PLUS Reagent Kit (Applied Biosystems, Waltham, MA, EE. UU.). Los datos transcriptómicos fueron normalizados mediante Robust Multichip Average (RMA), mientras que el efecto de lote fue corregido utilizando el algoritmo ComBat de la librería limma (lenguaje R)20. La matriz (genes x muestras) fue posteriormente colapsada mediante el algoritmo collapse_genes-09 (Fred's Softwares, Toulouse, Francia). Los genes diferencialmente expresados (GDE) se obtuvieron utilizando la librería de R, limma, con un valor p corregido (valor q) de 0.05, y siguiendo el flujo de trabajo recomendado20. Adicionalmente, los 5,000 genes más variables en el tejido CHC fueron identificados utilizando la desviación estándar. Los mapas de calor basados en la agrupación jerárquica no supervisada se generaron mediante la librería de R, pheatmap. La identificación de vías metabólicas de interés se realizó utilizando el algoritmo Sample Enrichment Score (SES) y las bases de datos KEGG, Reactome (reactome_2018803), Gene Ontology-Cellular Component (C5_CC) y MSigDB Hallmarks (H) compiladas por Fred's Softwares21. Los códigos fuente y las bases de datos de Fred's Software están disponibles en: https://sites.google.com/site/fredsoftwares/products.
Análisis del metilomaEl perfil de metilación de las muestras de CHC peruanos con código GSE136319 y GSE136380 fueron obtenidos a partir de la base de datos GEO. El metiloma de estas muestras se obtuvo utilizando los microarreglos e Infinium HumanMethylation450K MethylationEPIC BeadChip (Illumina, San Diego, CA, EE. UU.). Las posiciones diferencialmente metiladas (PDM) fueron identificadas mediante la librería R minfi utilizando un valor q a 0.0515. Los criterios considerados para el análisis de metilación fueron: Δβ promedio entre CHC y HNT > 5% (para las posiciones hipermetiladas) o≤5% (para las posiciones hipometiladas). Mediante el cálculo de la desviación estándar se identificaron igualmente las 5,000 posiciones con metilación más variable. La clasificación según asociación a «islas CpG» y «estructura genética» se realizó según lo definido por Illumina. Los mapas de calor basados en la agrupación jerárquica no supervisada se generaron mediante la librería de R, pheatmap. Para la identificación de vías metabólicas de interés se utilizó el algoritmo SES, y se utilizaron los genes que se encontraban asociados a las CpG de interés, según lo definido por Illumina.
Análisis estadísticosLos análisis de factor múltiple (AFM) y de componentes principales (ACP) se realizaron en R utilizando la librería FactoMineR22. Las variables con datos incompletos fueron tratados con la función imputeMFA de la librería R missMDA. Brevemente, como se recomienda en la guía de FactoMineR, se aplicó un AFM, puesto que nuestros datos contaban con variables numéricas y categóricas. El AFM fue realizado según las recomendaciones establecidas de la librería, y todos los grupos fueron considerados como variables relevantes, y ningún dato suplementario fue incorporado en el análisis. Adicionalmente, los t-test y χ2 fueron realizados en R. La significancia estadística fue evaluada uni o bilateralmente de acuerdo a las circunstancias. Todas las figuras del presente artículo fueron generadas en R y GraphPad Prism v8 (GraphPad Software, Boston, MA, EE. UU.).
Consideraciones éticasTodas las muestras incluidas en el presente estudio provienen de pacientes voluntarios, que aceptaron el uso de sus muestras y datos (en anonimidad) para investigación. El proyecto con código INEN 10-05, fue revisado y aprobado por el Comité Institucional de Ética en Investigación del INEN.
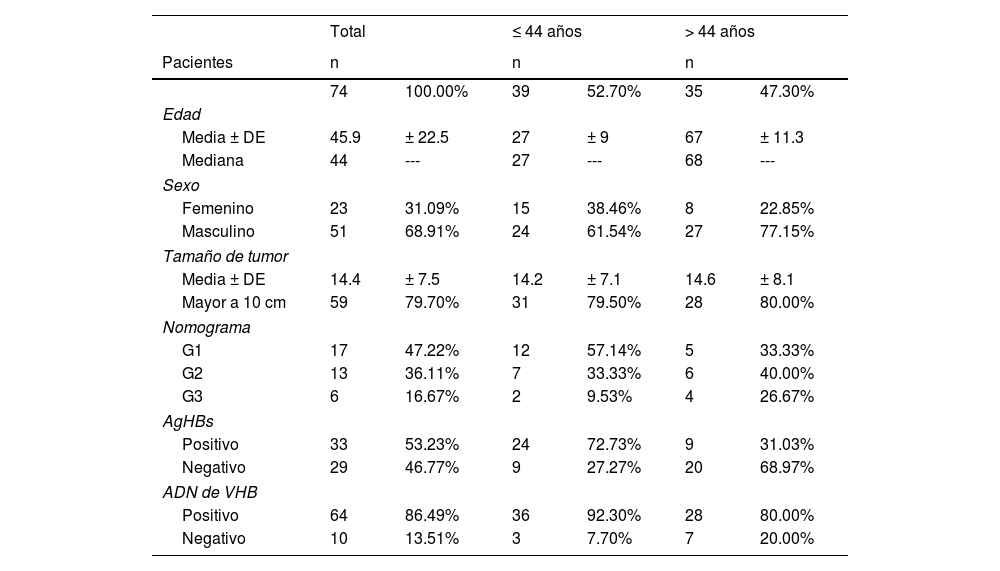
ResultadosVariables clínicas y demográficas asociadas a la edad en pacientes peruanos con CHCEn el presente estudio se incluyeron 74 pacientes peruanos con CHC tratados en el INEN por resección hepática. La edad promedio de estos fue de 45.9 años. De acuerdo a la mediana de la edad (44 años), los pacientes se clasificaron en adolescentes y jóvenes adultos (≤ 44, AJA), y en individuos de mediana o avanzada edad (> 44, MAE). El grupo AJA representa más de 50% (n = 39) de la población del estudio con CHC, con una media de 27 años, mientras que el grupo MAE (47.3%; n = 35) presenta una media de 67 años. La proporción según sexo (M/F) fue de dos hombres por cada mujer (2/1) para toda la cohorte de pacientes; sin embargo, en el grupo MAE, la proporción fue el doble (4/1). La media del diámetro tumoral fue de 14.4 cm (AJA, 14.2 cm y MAE, 14.6 cm; p > 0.05), y la gran mayoría de pacientes (∼80%) presentaron un tumor con un tamaño > 10 cm de diámetro. El nomograma de pronóstico de supervivencia para tumores > 10 cm18 pudo ser retrospectivamente aplicado en 36 pacientes (AJA = 21 y MAE = 15), 83% de los pacientes incluidos se encuentran en los grupos G1 (n = 17) y G2 (n = 13), ambos grupos presentan una mayor probabilidad de supervivencia en comparación con el G3 (n = 6), donde todos los pacientes fallecieron antes de cumplir los tres años posteriores a la operación (fig. 1a). El antígeno de superficie de hepatitis B (AgHBs) se detectó en 53% de pacientes (AJA = 72%, y MAE = 31%), mientras que el ADN del VHB se detectó en 86% de la población con CHC estudiada y el único subgenotipo identificado fue el F1b. Finalmente, el grupo AJA y MAE presentaron 92 y 80% (p > 0.05) de casos con presencia de ADN viral, respectivamente (tabla 1).
Evaluación de las variables clínicas y demográficas en pacientes peruanos con CHC. a) Curva de sobrevida de G1 (n = 17), G2 (n = 13) y G3 (n = 6), de 36 pacientes, clasificados según nomograma. Como resultado del análisis de AFM se muestra en un gráfico de componentes principales: b) la contribución de cada grupo de variables, y c) de las variables más relevantes dentro de cada grupo, para las dos dimensiones más relevantes (Dim. 1 y 2). Lab. (azul); Demog (verde y rojo); VHB (azul claro); Histo (amarillo y gris); Tumor (marrón y fucsia). Datos cualitativos (ql) y cuantitativos (qt). ACP de datos de d) expresión génica y e) metilación del ADN de pacientes peruanos representados según la infección por VHB, edad (AJA y MAE), y tipo de tejido (CHC y HNT).
Descripción clínica-patológica de la población con CHC incluida en el estudio
| Total | ≤ 44 años | > 44 años | ||||
|---|---|---|---|---|---|---|
| Pacientes | n | n | n | |||
| 74 | 100.00% | 39 | 52.70% | 35 | 47.30% | |
| Edad | ||||||
| Media ± DE | 45.9 | ± 22.5 | 27 | ± 9 | 67 | ± 11.3 |
| Mediana | 44 | --- | 27 | --- | 68 | --- |
| Sexo | ||||||
| Femenino | 23 | 31.09% | 15 | 38.46% | 8 | 22.85% |
| Masculino | 51 | 68.91% | 24 | 61.54% | 27 | 77.15% |
| Tamaño de tumor | ||||||
| Media ± DE | 14.4 | ± 7.5 | 14.2 | ± 7.1 | 14.6 | ± 8.1 |
| Mayor a 10 cm | 59 | 79.70% | 31 | 79.50% | 28 | 80.00% |
| Nomograma | ||||||
| G1 | 17 | 47.22% | 12 | 57.14% | 5 | 33.33% |
| G2 | 13 | 36.11% | 7 | 33.33% | 6 | 40.00% |
| G3 | 6 | 16.67% | 2 | 9.53% | 4 | 26.67% |
| AgHBs | ||||||
| Positivo | 33 | 53.23% | 24 | 72.73% | 9 | 31.03% |
| Negativo | 29 | 46.77% | 9 | 27.27% | 20 | 68.97% |
| ADN de VHB | ||||||
| Positivo | 64 | 86.49% | 36 | 92.30% | 28 | 80.00% |
| Negativo | 10 | 13.51% | 3 | 7.70% | 7 | 20.00% |
DE: desviación estándar; AgHBs: antígeno de superficie de hepatitis B; VHB: virus de hepatitis B.
Para el análisis de AFM, las variables fueron organizadas en ocho grupos, datos de laboratorio clínico (Lab.), demográficos cualitativos y cuantitativos (Demog ql y qt), asociados a la infección del VHB, histológicos cualitativos y cuantitativos (Histo ql y qt), y asociados al tumor cualitativos y cuantitativos (Tumor ql y qt) (Sup. Tabla 1). El análisis de AFM utilizando todos los datos clínicos y demográficos de los pacientes con CHC demostró que los grupos de variables Demog ql, Histo ql, Histo qt, Lab y Tumor qt, fueron los parámetros con mayor contribución (fig. 1b). Las variables con mayor relevancia dentro de estos grupos fueron, según el AFM, la edad, sobrevida, número de células mitóticas (mitosis), AFP, diámetro tumoral, bilirrubina total y albúmina (fig. 1c).
Adicionalmente, debido a la alta tasa de infección por el VHB, se realizó un ACP para determinar el efecto del VHB en la expresión y metilación. En ambos escenarios se observa una mayor proporción de pacientes AJA infectados con VHB, demostrando que el VHB actúa como un factor de confusión asociado a la edad (fig. 1d,e).
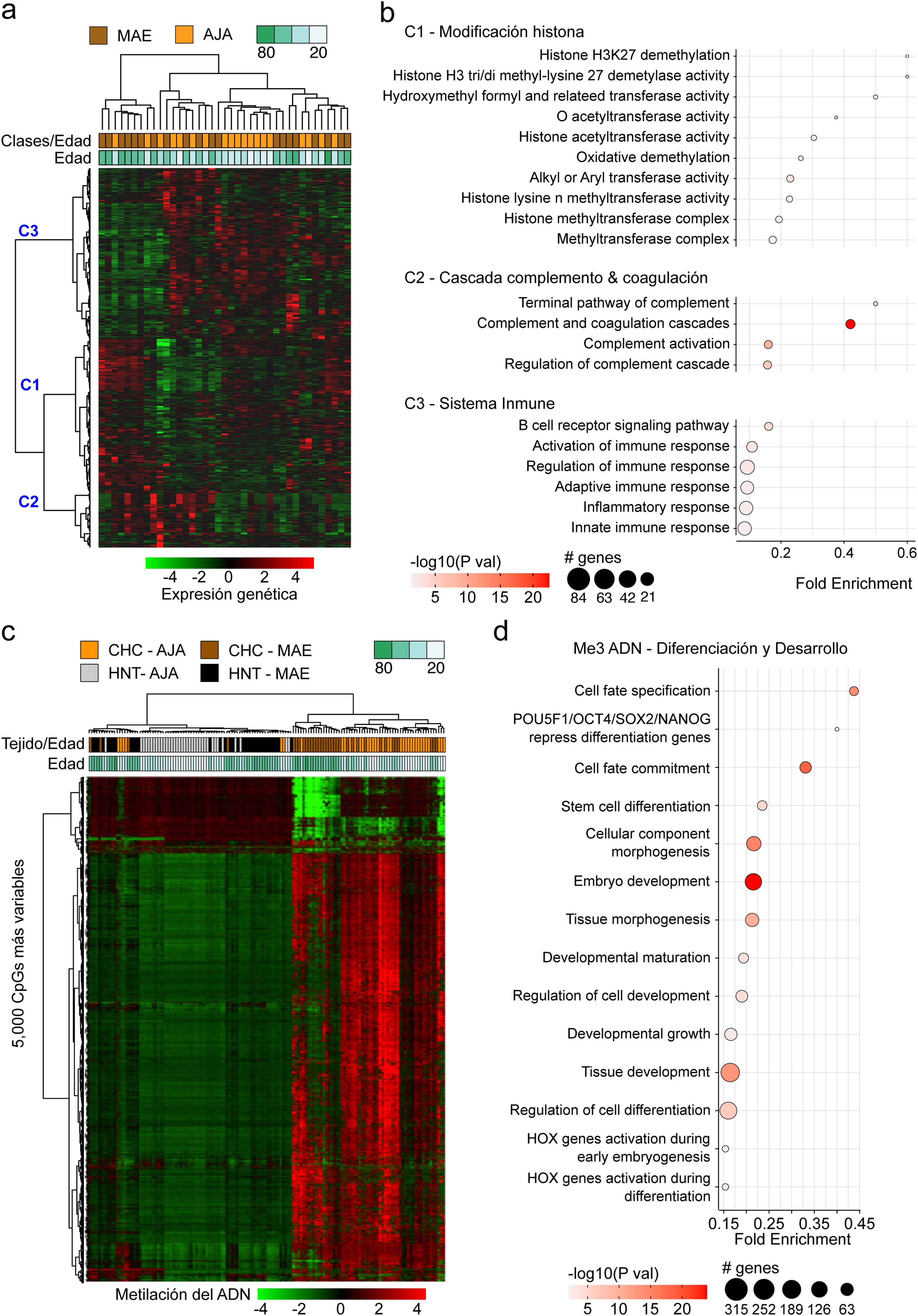
El perfil transcriptómico del CHC en Perú no se encuentra asociado a la edadEl perfil transcriptómico del CHC y HNT de un total de 39 pacientes (AJA = 19 y MAE = 20) se obtuvo a partir de datos previamente publicados15. La evaluación de los 5,000 genes más variables en el CHC demostró que no existe diferencia significativa entre los dos grupos etarios (p > 0.05) (fig. 2a). Sin embargo, un análisis de agrupamiento jerárquico no supervisado de los 5,000 genes más variables en el tejido CHC permitió evidenciar que estos se dividían en tres grandes grupos (C1-C3). Los genes que se encontraban en C1 estaban principalmente asociados con la regulación de la expresión mediante la modificación de histonas, mientras que en C2 la mayoría participaban en la cascada del complemento y la coagulación, y en C3 se encontraban genes que participan en la respuesta inmune. Otras vías menos representativas fueron la regulación de hormonas (C1), metabolismo del retinol y lípidos (C1), así como la reparación del ADN (C2) (fig. 2b).
Análisis del perfil transcriptómico y de metilación del ADN de pacientes peruanos con CHC. Se identificaron los 5,000 genes (a,b) y 5,000 CpG más variables (c,d). Utilizando (a) los niveles de expresión (AJA = 19, MAE = 20) y (c) de metilación del ADN (AJA = 39, MAE = 35) de estos se generaron mapas de calor basados en una agrupación jerárquica no-supervisada. En (a) se destacan tres grandes grupos de genes C1, C2 y C3. Las vías con mayor valor de significancia y de fold enrichment (b) de los 5,000 genes, y d) de las 5,000 CpG más variables se representaron en un gráfico de dispersión. El fold enrichment es la proporción de genes de la lista de interés que se encuentra en la vía evaluada.
El perfil de metilación del CHC y HNT de 70 pacientes (AJA = 36 y MAE = 34) se obtuvieron de un estudio previo15. Similar al análisis transcriptómico, las 5,000 CpG más variables fueron identificadas, y se realizó un agrupamiento jerárquico no supervisado. En contraste a su contraparte no-tumoral (HNT), el CHC peruano muestra un aumento global de metilación del ADN (hipermetilación), con mayor intensidad en los pacientes jóvenes (fig. 2c). Además, estas CpG están principalmente asociadas a genes implicados en la diferenciación celular, con marcas de metilación del residuo de lisina en la posición N-terminal 27 de la proteína histona H3 [H3K27 (me3)], y blancos del complejo represivo polycomb 2 (PRC2) (fig. 2d).
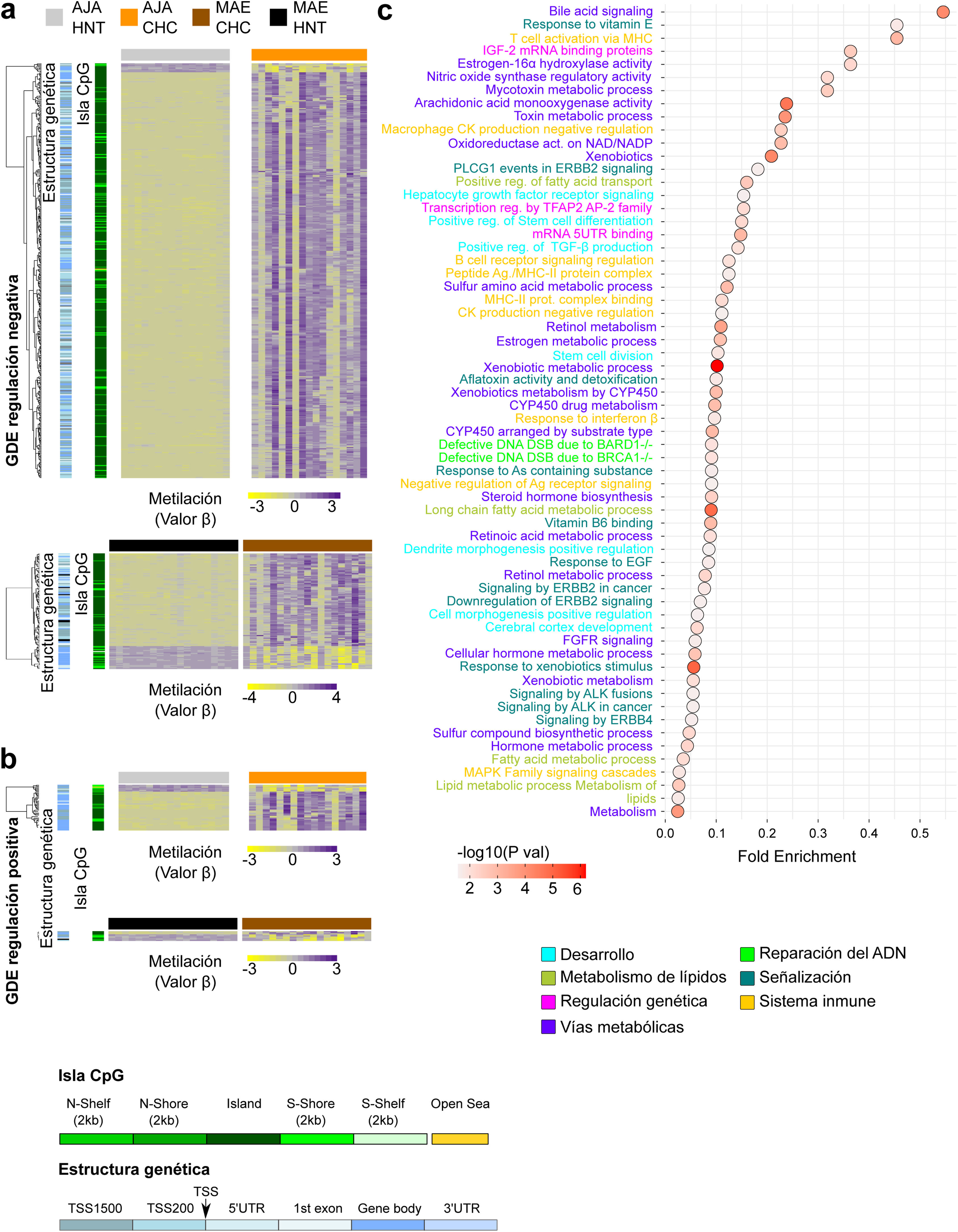
La integración del perfil transcriptómico y metilación resalta procesos de diferenciación celular y del sistema inmune en pacientes de CHC peruanosPara una mejor comprensión de la metilación del ADN de las CpG se realizó un mapa de calor sobre los genes regulados positiva y negativamente15. Se consideraron de forma independiente las regiones que abarcan la estructura genética y la isla CpG. Las CpG asociadas a genes regulados negativamente (en su mayoría hipermetilados) se detectaron principalmente en islas CpG dentro de las regiones promotoras de TSS200 y TSS1500; mientras que las CpG asociadas a genes regulados positivamente (en su mayoría hipometilados) se ubicaron en regiones no relacionadas a islas CpG en el cuerpo del gen (fig. 3a,b). La integración genómica en CHC también reveló un predominio de vías relacionadas al metabolismo celular, sin embargo, también resaltan vías relacionadas a procesos de diferenciación celular y del sistema inmune.
Análisis integrado del perfil transcriptómico y de metilación del ADN de pacientes peruanos con CHC. Se utilizaron mapas de calor basado en una agrupación jerárquica no-supervisada para mostrar los niveles de metilación del ADN (valor β) de (a) los genes diferencialmente expresados (GDE) regulados negativamente y (b) positivamente, en AJA (n = 39, panel superior) y MAE (n = 35, panel inferior). a,b) El dendrograma izquierdo muestra la anotación de la isla CpG y estructura del gen. c) Las vías con mayor valor de significancia de fold enrichment asociados al análisis integrado de la expresión de génica y metilación del ADN, se ilustraron utilizando un gráfico de dispersión. El fold enrichment es la proporción de genes de la lista de interés que se encuentra en la vía evaluada.
Finalmente, se exploraron las diferencias entre los grupos definidos según el nomograma de pronóstico de supervivencia para tumores con un tamaño > 10 cm, a nivel transcriptómico y de metilación del ADN. Únicamente 36 pacientes (perfil transcriptómico = 17 y perfil de metilación del ADN = 36) contaban con las variables preoperativas necesarias para poder ser evaluados. Un total de 17 pacientes obtuvieron 0 puntos, por lo tanto fueron clasificados en G1, mientras que 13 obtuvieron entre 1-100 puntos y seis muestras > 100, siendo clasificadas en los grupos G2 y G3, respectivamente (tabla 1). Según esta clasificación, 47% de pacientes tienen una alta probabilidad de sobrevida a largo plazo, mayormente ≤ 44 años (AJA = 12; MAE = 5), 36% una alta probabilidad de recurrencia durante los primeros 12 meses (AJA = 7, MAE = 6), y 17% alta probabilidad de muerte postoperatoria (AJA = 2, MAE = 4). A pesar de estas diferencias clínicas, los análisis no demostraron diferencias significativas a nivel transcriptómico, ni de metilación del ADN entre los tres grupos. Por esta razón no se profundizó más con estos análisis. Sin embargo, se observó que más de 50% de pacientes ≤ 44 años (57%) tienen una alta probabilidad de sobrevida a largo plazo.
DiscusiónEn el presente estudio describimos la relación entre las características clínico-patológicas y un perfil molecular divergente de CHC que no ha sido reportado anteriormente en otras partes del mundo. La presencia de infecciones ocultas del VHB (IOB) incrementan la frecuencia de VHB en los casos de CHC en la población peruana, siendo el principal factor de riesgo para el desarrollo de CHC. Aunque la bimodalidad asociada a la edad podría sugerir características particulares para cada grupo etario (AJA y MAE), no se evidenció en el perfil transcriptómico. Sin embargo, la hipermetilación global del ADN en la población peruana con CHC se observó con mayor intensidad en AJA. Este fuerte patrón de hipermetilación en pacientes andinos con CHC sería el perfil característico del CHC en el Perú.
Estudios previos de nuestro grupo de investigación han caracterizado en diferentes áreas de estudio y de forma sistémica el CHC en la población peruana. El conjunto de estos hallazgos ha demostrado que en una fracción significativa de CHC en la población peruana presenta características únicas no reportadas en otras poblaciones12,18,19,23,24.
Como era de esperarse, se observó bimodalidad equilibrada entre AJA y MAE, lo que coincide con previas cohortes de CHC analizadas en Perú12,13, por otro lado, la distribución por género se mantiene equilibrada en comparación con otras regiones2. El CHC en AJA es generalmente de subtipo fibrolamelar y considerado como un tipo raro de presentación en algunas regiones, especialmente en Asia25. Sin embargo, en las Américas los casos de CHC en AJA son más comunes y mayormente asociados a una infección con VHB, sin presentar características del subtipo fibrolamelar14,26,27. No obstante, en el Perú se observa un porcentaje muy superior a lo reportado en otras regiones de las Américas, en más de 85% de casos totales de CHC se ha encontrado la presencia de ADN viral (VHB), mientras que en AJA fue en más de 90%24,28.
Aunque se sabe que las altas cargas virales de una infección crónica por el VHB es una de las principales causas de la hepatocarcinogénesis29, estudios en la población peruana con CHC han demostrado una carga viral baja, que podría funcionar en sinergia ante la presencia de otros factores tumorigénicos19. Adicionalmente, este estudio ha reportado una gran cantidad (> 40%) de IOB en tejido tumoral (CHC) y no tumoral (HNT), detectada únicamente por métodos moleculares ultrasensibles (como la droplet digital PCR)19,26. La mayoría de las muestras con IOB dieron resultados negativos para AgHBs, por lo que existe la necesidad de implementar métodos más sensibles para la detección del VHB en el Perú. Siendo la infección del VHB un factor asociado al CHC, conocer la verdadera prevalencia del VHB en la población peruana permitiría entender mejor su rol en el desarrollo de esta enfermedad.
Otra particularidad del CHC peruano es su gran tamaño al momento del diagnóstico, aproximadamente 80% de pacientes presenta un tumor con más de 10 cm de diámetro, cómo se ha reportado anteriormente13,14,16. Debido a la falta de algoritmos clínicos que ayuden a predecir la supervivencia de estos pacientes luego de una resección hepática, en el 2021 Ruiz et al. reportaron un nomograma capaz de determinar la sobrevida en pacientes con tumores gigantescos18. Utilizando este algoritmo, se determinó el grupo de sobrevida de 36 pacientes (tabla 1), y con el fin de encontrar una firma molecular característica para cada grupo, se realizó una comparación de sus perfiles de expresión y metilación del ADN. Lamentablemente, no se encontraron diferencias significativas entre los tres grupos de sobrevida, mostrando que el CHC en la población peruana es una entidad única o con poca variabilidad molecular15.
En el presente estudio no se observaron diferencias significativas en el perfil transcriptómico de AJA y MAE, incluso aplicando otro método de análisis, como se ha descrito anteriormente15. Los análisis de enriquecimiento permitieron identificar grupos de vías relacionadas a la regulación de la expresión, vía la modificación de la lisina 27 en la histona 3 (H3K27). La modificación de H3K27 es catalizada por un complejo de modificación enzimática, conocido como PRC2. Por lo que estos resultados van de acuerdo con lo descrito por Cerapio et al., quienes demostraron que PRC2 podría ser uno de los principales actores moleculares de la reprogramación de las células cancerígenas del CHC en pacientes peruanos15. Demostrando que los mecanismos moleculares responsables de la carcinogénesis en este nuevo subtipo de CHC son sin precedentes en la literatura.
La evaluación del perfil de metilación confirma lo reportado anteriormente en pacientes peruanos15, un fenotipo de hipermetilación del ADN. Estos resultados difieren de lo reportado en otras regiones, donde el CHC presenta un perfil de hipometilación global, aunque la literatura describe islotes de hipermetilación focal en genes conductores9,10. El perfil de hipermetilación global observado en el CHC peruano se ha reportado únicamente en células madre pluripotentes humanas inducidas (iPSCs) con capacidad de reprogramación celular30. Este fenotipo biológico estaría de acuerdo con lo descrito por Cerapio et al. y con lo observado en este trabajo, que las 5,000 CpG más variables se encontraban asociadas a genes involucrados en la diferenciación y desarrollo celular15.
La integración de los perfiles de transcripción y metilación del ADN en CHC peruano evidencia una disminución en la expresión de genes probablemente debido a la hipermetilación del ADN, perfil similar a células embrionarias15,30. Sin embargo, para corroborar esta hipótesis, deben desarrollarse estudios comparativos con células en procesos de diferenciación para contrastar con el perfil molecular del CHC en la población peruana.
En conclusión, el presente estudio muestra un CHC con características únicas que no ha sido previamente reportado en otras regiones. Las infecciones con VHB son altamente prevalentes y podrían permanecer ocultas a los métodos de diagnóstico de rutina, por lo que métodos más sensibles para el diagnóstico deben ser implementados. La integración de datos del perfil de transcriptoma y metilación del ADN direccionan futuras investigaciones encaminadas a explorar en profundidad el perfil de metilación de CHC con características de células indiferenciadas o en proceso de diferenciación.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
FinanciamientoLos autores declaran que este trabajo fue apoyado por el ITMO Cáncer de la Alianza Nacional Francesa para las Ciencias de la Vida y la Salud (Aviesan) y del Instituto Nacional francés del Cáncer (INCa) sobre fondos administrados por el Instituto Nacional francés de Salud e Investigación Médica (Inserm) (subvención 21CD025-00), así como por el Programa de Laboratorios Mixtos Internationales (LMI) del Instituto francés de Investigación para el Desarrollo (IRD). JCM reconoce el apoyo financiero del Proyecto Concytec-Banco Mundial «Mejoramiento y Ampliación de los Servicios del Sistema Nacional de Ciencia Tecnología e Innovación Tecnológica» 8682-PE, a través de su unidad ejecutora ProCiencia [08-2018-FONDECYT/BM-Programas de Doctorados en Áreas Estratégicas y Generales]. JPC fue beneficiario de una beca posdoctoral de la Agencia Nacional de Investigación de Francia (ANR) en el marco del Programa de Laboratorios de Excelencia 11-LABX-0068.