El cáncer de estómago (CE) y colorrectal (CCR) presentan altas tasas de incidencia y mortalidad en la población mundial. Estas 2 neoplasias se caracterizan por tener una gran heterogeneidad genética. Hasta el momento, no existen estudios moleculares que analicen las mutaciones en los genes APC, KRAS y TP53 en población colombiana/latinoamericana.
ObjetivoAnalizar mutaciones en los genes APC, KRAS y TP53 en 59 pacientes con CE y CCR mediante el secuenciamiento directo.
Pacientes y métodosSe estudió a 29 pacientes con CE y 30 con CCR. Se realizó un análisis de mutaciones en los 3 genes por las técnicas de reacción en cadena de la polimerasa y secuenciamiento directo.
ResultadosSe encontró una frecuencia total de mutaciones del 30.5%. El gen más frecuentemente mutado fue APC (15.3%), seguido de KRAS (10.1%) y TP53 (5.1%). Las muestras de CCR presentaron una frecuencia de mutaciones del 46,7% y las CE del 13.3% (p=0.006). No se encontraron mutaciones simultáneas en los 3 genes. En solo 6 muestras de tumores (10%) se detectaron mutaciones en 2 genes. Adicionalmente, se obtuvo una alta frecuencia de polimorfismos en ambos tipos de cáncer, el más común fue el rs41115 localizado en el gen APC.
ConclusiónLas mutaciones en los genes APC, KRAS y TP53 fueron más comunes en el CCR que en el CE; nuestros resultados indican la existencia de diferentes vías genéticas en la carcinogénesis del CE y del CCR, y revelan una frecuencia de mutaciones particular en los pacientes colombianos estudiados, que podría estar influida por factores ambientales y étnicos, y el estilo de vida de esta población.
Stomach cancer (SC) and colorectal cancer (CRC) present with high rates of incidence and mortality in the worldwide population. These 2 tumors are characterized by great genetic heterogeneity. Up to now, there have been no molecular studies that analyze the mutations in the APC, KRAS, and TP53 genes in the Colombian/Latin American population.
ObjectivesTo analyze mutations in the APC, KRAS, and TP53 genes through direct sequencing in 59 patients with SC and CRC.
Patients and methodsTwenty-nine patients with SC and 30 with CRC were studied. An analysis of the mutations of the 3 genes was carried out using polymerase chain reaction and direct sequencing techniques.
ResultsA 30.5% total mutation frequency was found. The most frequently mutated gene was APC (15.3%), followed by KRAS (10.1%) and TP53 (5.1%). The CRC samples had a mutation frequency of 46.7% and it was 13.3% in the SC samples (P=.006). No mutations occurred simultaneously in the 3 genes. Mutations in 2 genes were found in only 6 tumor samples (10%). There was also a high frequency of polymorphisms in both types of cancer, the most common of which was the rs41115 polymorphism, located on the APC gene.
ConclusionThe APC, KRAS, and TP53 gene mutations were more common in CRC than in SC. Our results suggest the existence of different genetic pathways in the carcinogenesis of SC and CRC and they also reveal a particular mutation frequency in the Colombian patients studied; this could be influenced by factors related to the environment, ethnicity, and lifestyle of this population.
Los cánceres de estómago (CE) y colorrectal (CCR) presentan altas tasas de incidencia y mortalidad en la población mundial1-3. La situación es más preocupante en países en vía de desarrollo, porque la mortalidad para ambas malignidades tiene una tendencia al aumento y en la mayoría de los casos son diagnosticados en estadios avanzados con un mal pronóstico4. El CE es la cuarta causa de cáncer y la segunda causa de muerte por cáncer en el mundo; es más frecuente en hombre que en mujeres1. La incidencia del CE varía geográficamente, con una alta incidencia en países asiáticos, como Corea, Japón y China, y en algunos países de Latinoamérica1. En Colombia, el CE ocupa el segundo lugar en incidencia en hombres y mujeres, y es la primera causa de muerte por cáncer1. Más del 90% de los casos de CE son adenocarcinomas y se clasifican histológicamente en 2 tipos: difuso e intestinal5,6. La patogénesis del CE ocurre por múltiples factores etiológicos, que incluyen los genéticos, ambientales, dietarios, estilo de vida y la infección con la bacteria Helicobacter pylori (H. pylori)7-9.
Por otra parte, el CCR es la tercera neoplasia más frecuente en hombres y mujeres en el mundo; los países desarrollados presentan las mayores tasas de incidencia1. En Colombia, el CCR, en ambos sexos, ocupa el sexto puesto en incidencia y el cuarto en mortalidad1. Similar a lo observado en el CE, el CCR presenta una distribución geográfica variable, diversos factores están involucrados en la etiología del CCR, como son los genéticos, los ambientales, la raza y el estilo de vida, entre otros10. La mayoría de los casos de CCR son esporádicos y un bajo porcentaje se relaciona con la historia familiar11,12.
En Colombia, el CE y el CCR presentan una distribución geográfica variable, con altas incidencias en la región central, noroeste, sur occidente, oriente y sur del país, mientras que bajas incidencias se observan en las regiones del Caribe (norte) y pacífica del país. Esta disparidad podría deberse a los hábitos en la dieta y a las preparaciones caseras de alimentos con alto contenido de nitritos típicos de cada región, así como a una alta ingesta de carnes rojas ahumadas, alcohol y al tabaquismo6.
El CE y el CCR son enfermedades muy heterogéneas que se originan por diversas vías genéticas; son comunes las alteraciones cromosómicas, las mutaciones en oncogenes, los genes supresores de tumores, los genes de reparación y la metilación en genes que controlan el ciclo celular13-15. Particularmente en el CCR, Fearon y Vogelstein propusieron un modelo molecular que describe la secuencia de la carcinogénesis colorrectal a partir de adenoma a carcinoma y en el que ocurren mutaciones en diversos oncogenes y genes supresores de tumores14.
De otro lado, estudios genéticos en CE y CCR reportan que alteraciones en los genes APC, KRAS, TP53, CDH1, MLH1 y ERBB2 están entre los cambios moleculares más comunes, adquiridos en la carcinogénesis de estas 2 malignidades2,16-20. Por lo anterior, se propone que estos genes son esenciales para la transformación de las células normales hacia carcinoma. De esta manera, la caracterización molecular de las vías genéticas del CE y CCR es de gran importancia para su posterior correlación con las características clínico-patológicas y con el pronóstico de los pacientes21-23.
En este trabajo, se formuló la hipótesis de que la frecuencia de mutaciones en los genes APC, KRAS y TP53 en las muestras de CE y CCR de pacientes colombianos es diferente de la de otras poblaciones.
El objetivo de nuestro estudio fue analizar las mutaciones somáticas en los genes APC, KRAS y TP53 en pacientes colombianos con cáncer de estómago y colorrectal, mediante el secuenciamiento directo.
Materiales y métodosPoblación y muestraSe realizó un estudio descriptivo, de corte transversal. La población de estudio estuvo constituida por 59 pacientes, 29 con CE y 30 con CCR, todos de tipo esporádico. El estudio incluyó a 27 mujeres y 32 hombres. Ninguno de los pacientes tenía antecedentes de cáncer y no recibieron tratamiento antineoplásico previo a la cirugía. Todos los casos de CE y CCR fueron confirmados por el estudio histopatológico. A los pacientes se les solicitó la participación voluntaria en el estudio y la firma del consentimiento informado. El protocolo del estudio fue aprobado por el Comité de Bioética para experimentación en humanos de la Universidad de Antioquia, Medellín, Colombia.
Las muestras de los tejidos tumorales primarios fueron obtenidos por los cirujanos mediante resección quirúrgica en 2 instituciones de la ciudad de Medellín, Colombia: el Hospital Universitario San Vicente Fundación y la Clínica León XIII, durante los años 2010 al 2011. Cada una de las muestras tumorales se dividieron en 2 porciones, una para el estudio histopatológico, realizado por un patólogo experto, y la otra porción se almacenó a –80°C para el posterior análisis genético.
Extracción de ADNLa extracción del ADN del tejido tumoral primario se realizó utilizando el kit comercial QIAamp DNA Mini Kit (Qiagen, Hilden, Alemania), siguiendo las recomendaciones de la casa comercial. Posteriormente, el ADN se cuantificó en un espectrofotómetro NanoDrop 2000c Spectrophotometer (Thermo Scientific, EE. UU.) y se determinó su integridad mediante una electroforesis en gel de agarosa al 1%. El ADN extraído se almacenó a –20°C.
Amplificación del ADN y análisis de mutaciones en los genes APC, KRAS y TP53El ADN aislado se amplificó por el método de la reacción en cadena de la polimerasa (PCR) en un termociclador Gene Amp PCR System 9700 (Applied Biosystems, EE. UU.), utilizando cebadores específicos para cada gen; en el gen APC se amplificó la región «Mutation Cluster Region» (MCR) ubicada en el exón 15; para del gen KRAS se amplificó el exón 2 (codones 12 y 13) y los exones 5 al 8 de TP53. Brevemente, la PCR se realizó en un volumen de 35μl que contenía 300 ng del ADN tumoral en una mezcla de reacción con las siguientes concentraciones: 1X para el buffer de reacción 10X, 200μM de dNTPs, 0.4μM de cada primer y 1.4 U de Taq ADN polimerasa (Invitrogen, EE. UU.). Las concentraciones de MgCl2 fueron 1.05mM para APC y KRAS, y 1.5mM para TP53. Los productos amplificados por PCR se examinaron mediante electroforesis en geles de agarosa al 2%, se colorearon con bromuro de etidio y se almacenaron a –20°C hasta el momento de realizar el secuenciamiento.
SecuenciamientoTodas las muestras amplificadas se purificaron y secuenciaron directamente por ambas cadenas. El secuenciamiento se realizó en un analizador genético automático 3730xl DNA Analyzer de Applied Biosystems.
Análisis de resultadosLos cromatogramas obtenidos se editaron con el programa Chromas Pro y se alinearon con las secuencias de referencia publicadas en el GenBank (NCBI), cuyos códigos de acceso son: NT_034772.6 (APC), NT_009714.17 (KRAS) y NT_010718.16 (TP53).
Análisis estadísticoLos resultados se analizaron mediante el programa SPSS versión 18. En la estadística descriptiva para variables cuantitativas se utilizaron la media y el rango; las variables categóricas se presentaron en frecuencias y porcentajes. Para la comparación de variables categóricas, como género y frecuencia de mutación, se realizó la prueba de la χ2 de Pearson, cuando los valores esperados eran inferiores a 5 se utilizó la corrección de Yates. La comparación entre las variables cuantitativas continuas como la edad se realizó mediante la prueba de la t de Student de muestras independientes. Todos los valores de p calculados son bilaterales y se consideró significativo un valor p<0.05.
ResultadosSe estudió a un total de 59 pacientes, con una media de edad de 64.1 años (rango 12-94). Los pacientes con CCR y CE fueron estratificados por edad y género, para el primer grupo la edad promedio fue 62,1 años (rango 12-84) y para el segundo grupo fue de 66,1 años (rango 36-94); no se observó diferencia entre ambos grupos (p=0.332). Con relación al género, el 63.3% de los pacientes con CCR eran mujeres y el 36.7 eran hombres, mientras que en el CE el 27.6% eran mujeres y el 72.4% eran hombres, con una diferencia entre los 2 grupos estadísticamente significativa (p=0.005).
Entre los síntomas y signos más comunes que presentaron los pacientes con CE se encuentran: epigastralgia, emesis, astenia, adinamia, anorexia, nauseas, anemia, sangrado, pérdida de peso y obstrucción. En cuanto a los síntomas de los pacientes con CCR, los más frecuentes fueron: sangrado rectal, pérdida de peso, obstrucción del colon, estreñimiento, dolor abdominal difuso, melenas y distensión abdominal.
La frecuencia total de mutaciones en las 59 muestras analizadas fue del 30.5% (18/59) (tabla 1). Adicionalmente, se encontró una alta frecuencia de polimorfismos en los 3 genes; en total, se identificaron 7 polimorfismos diferentes en las 59 muestras; el más frecuente fue el rs41115 (c.4479G>A, p.Thr1493Thr) ubicado en el exón 15 del gen APC. Todos los polimorfismos identificados en este estudio están reportados en la base de datos de SNP24.
Mutaciones identificadas en los genes APC, KRAS y TP53 en las muestras de CE y CCR analizadas
| Tumor | Gen | Caso N.° | Exón | Codón | Posición | Mutación | Cambio de aminoácido | % mutación |
| Estómago | APC | 08 | 15 | 1465 | c.4393_4394 delAG | AGAGAG→AGAG | p.Ser1465TrpfsX3 | 3.3 |
| KRAS | 04 | 2 | 12 | c.35G>A | GGT→GAT | p.Gly12Asp | 6.7 | |
| 08 | 2 | 13 | c.38G>A | GGC→GAC | p.Gly13Asp | |||
| TP53 | 017 | 5 | 173 | c.517G>A | GTG→ATG | p.Val173Met | 3.5 | |
| Colorrectal | APC | 031 | 15 | 1465 | c.4393_4394dupAG | AGAG→AGAGAG | p.Ser1465ArgfsX9 | 26.7 |
| 032 | 15 | 1367 | c.4099C>T | CAG→TAG | p.Gln1367X | |||
| 037, 039 | 15 | 1556 | c.4666dupA | AAC→AAAC | p.Thr1556AsnfsX3 | |||
| 38 | 15 | 1443 | c.4329T>A | CCA→CCT | p.Pro1443Pro | |||
| 15 | 1474 | c.4420A>G | ACT→GCT | p. Ala1474Thr | ||||
| 052 | 15 | 1306 | c.3916G>T | GAA→TAA | p.Glu1306X | |||
| 053 | 15 | 1309 | c.3927_3931delAAAGA | AAAAGAAAAGA→AAAAGA | p.Glu1309AspfsX4 | |||
| KRAS | 031, 037 | 2 | 12 | c.35G>A | GGT→GAT | p.Gly12Asp | 13.3 | |
| 033 | 2 | 12 | c.35G>T | GGT→GTT | p.Gly12Val | |||
| 053 | 2 | 13 | c.38G>A | GGC→GAC | p.Gly13Asp | |||
| TP53 | 032 | 6 | 188 | c.562C>T c.563T>A | CTG→TAG | p.Leu188X | 6.7 | |
| 039 | 6 | 199 | c.595G>T | GGA→TGA | p.Gly199X |
Al comparar la frecuencia de mutaciones de APC entre el CE y CCR (p=0.006).
Al comparar la frecuencia de mutaciones de KRAS entre el CE y CCR (p=0.06).
Al comparar la frecuencia de mutaciones de TP53 entre el CE y CCR (p=0.98).
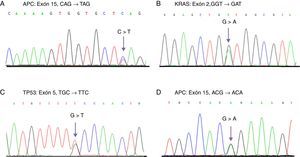
En las muestras de CE, se encontró una sola mutación en el gen APC (3.5%); esta mutación fue una deleción de 2 nucleótidos (AG) en la posición c.4393_4394delAG, la cual genera un codón de parada prematuro en la proteína (p.Ser1465ArgfsX9) (tabla 1). En estas mismas muestras, también se identificó el polimorfismo rs41115 con una alta frecuencia del 86.2% (25/29) (tabla 2) (fig. 1). Por el contrario, en las muestras de CCR, la frecuencia de mutaciones identificadas en APC fue del 26.6% (8/30). Cuatro de estas mutaciones son de tipo de cambio en el marco de lectura y las otras 4 fueron del tipo de sustitución de bases; en la tabla 1 se describen con mayor información las mutaciones detectadas (fig. 1). Al comparar la frecuencia de mutaciones en APC, se encontró una tendencia a una mayor frecuencia en CCR que en CE (p=0.06). Por otro lado, en estas muestras se identificaron 2 polimorfismos, el rs41115 con una frecuencia del 56.7% (17/30) y el rs1801166 (c.3949G>C, p.Glu1317Gln) con el 3.3% (1/30) (tabla 2).
Polimorfismos identificados en los genes APC, KRAS y TP53 en las 59 muestras de pacientes con CE y CCR evaluados
| Gen | Exón/intrón | Código SNP | Cambio en la proteína | cADN | CCR | CE | p | ||
| n | % | n | % | ||||||
| APC | Exón 15 | rs41115 | p.T1493T | c.4479G>A | 18 | 60 | 25 | 86.2 | 0.008a |
| rs1801166 | p.Glu1317Gln | c.3949G>C | 1 | 3.3 | – | – | |||
| KRAS | Intrón 2 | rs61759635 | – | c.111+116_111+120delAGTTA | 1 | 3.3 | 1 | 3.5 | 0.4b |
| rs12228277 | – | c.111+190A>T | 11 | 37 | 8 | 27.6 | |||
| TP53 | Exón 6 | rs1800372 | p.Arg213Arg | c.639A>G | – | – | 1 | 3.5 | 0.69c |
| Intrón 7 | rs12947788 | – | c.782+72C>T | 5 | 17 | 5 | 17.2 | ||
| rs12951053 | – | c.782+92T>G | 5 | 17 | 5 | 17.2 | |||
| Total | 41 | – | 45 | – | |||||
CCR: cáncer colorrectal; CE: cáncer de estómago; SNP: single nucleotide polymorphism.
Cromatogramas obtenidos del secuenciamiento directo que muestra diferentes mutaciones identificadas. A) Mutación en APC detectada en una muestra de CCR. B) Mutación en KRAS identificada en muestras de CE y CCR. C) Mutación en TP53 encontrada en un caso de CE. D) Polimorfismo p.T1493T en APC detectado con una alta frecuencia en pacientes con CE y CCR. Las flechas muestran el sitio del cambio de base.
Por otra parte, la frecuencia de mutaciones del gen KRAS en las muestras de CE fue del 6.9% (2/29) (tabla 1). Las mutaciones fueron 2 sustituciones de bases que ocurrieron en los codones 12 y 13; ambas mutaciones generan un cambio en la secuencia de la proteína: c.35G>A (p.Gly12Asp) y c.38G>A (p.Gly13Asp) (tabla 1, fig. 1). Además, se identificaron 2 polimorfismos en el intrón 2; el más frecuente fue el rs12228277 (c.111+190 A>T), en el 27.6% de las muestras (8/29) (tabla 2). El otro polimorfismo fue el rs61759635 (c.111+116_111+120delTAACT); se identificó en una sola muestra (3.5%) y consistió de una deleción de 5 nucleótidos. La frecuencia de mutaciones de KRAS en las muestras del CCR fue del 13.3% (4/30) (tabla 1); todas las mutaciones detectadas fueron sustituciones de base, 3 en el codón 12 y una en el codón 13 (tabla 1). Al comparar la frecuencia de mutaciones en KRAS entre el CE y el CCR, no se encontraron diferencias (p=0.69). Al igual que en el análisis genético de KRAS en las muestras de CE, en el CCR se identificaron 2 polimorfismos con una alta frecuencia, del 40% (12/30); el polimorfismo más frecuente fue el rs12228277 con el 36.7% (11/30), mientras que el polimorfismo rs61759635 se presentó en el 3.3% (1/30) (tabla 2).
Por último, en las muestras de CE se encontró una frecuencia de mutación para el gen TP53 del 3.5% (1/29) (fig. 1). La mutación c.517G>A (p.Val173Met) se detectó en el exón 5 (tabla 1). Además, se identificaron 3 polimorfismos: uno en el exón 6, rs1800372 (c.639A>G, p.Arg213Arg) en el 3.5% y 2 polimorfismos, rs12947788 (c.782+72C>T) y rs12951053 (c.782+92T>G) en el intrón 7 que cosegregan juntos, con una frecuencia del 17.2% (5/29) (tabla 2). De forma similar a lo encontrado en las muestras de CE, en las de CCR se identificaron 2 (6.6%) mutaciones en este gen (p=0.98); ambas mutaciones fueron sustituciones de bases en el exón 6 y generaron un codón de parada prematuro (tabla 1). Nuevamente, se identificaron 2 polimorfismos en el intrón 7 en el 16.7% (5/30) de los casos (tabla 2).
Con relación a la histología de los casos de CE, se observó que el tipo intestinal fue el más común, con el 48.3% (14/29), seguido del difuso, 41.4% (12/29); se encontró que la mitad de los casos presentaban un estadio avanzado del cáncer (iii y iv), con mayor proporción en el estadio iii. De este grupo de pacientes, el 20.7% tenía metástasis. Las 4 mutaciones identificadas en CE se presentaron en los adenocarcinomas de tipo intestinal, los cuales tenían un estado avanzado de malignidad (estadio iv). La localización de los casos de CE fue principalmente en el antro, el 58.6% (17/29) (tabla 3).
Descripción general de las características clínico-patológicas y de las frecuencias de mutaciones en los 29 pacientes con CE analizados
| Total, n (%) | APC | KRAS | TP53 | ||||
| Normal, n (%) | Mutado, n (%) | Normal, n (%) | Mutado, n (%) | Normal, n (%) | Mutado, n (%) | ||
| Número de pacientes | 29 | 28 (96.6) | 1 (3.4) | 28 (93.3) | 2 (6.7) | 28 (93.3) | 2 (6.7) |
| Edad (media) | 65.5 | 66.1 | 64 | 67.8 | 70 | 66.5 | 65 |
| Género | |||||||
| Femenino | 8 (27.5) | 7 (24.1) | 1 (3.4) | 7 (24.1) | 1 (3.4) | 8 (26.7) | – |
| Masculino | 21 (72.4) | 21 (72.4) | – | 20 (69) | 1 (3.4) | 20 (69) | 1 (3.4) |
| Estadio del CE | |||||||
| I | 2 (6.9) | 2 (6.9) | – | 2 (6.9) | – | 2 (6.9) | – |
| II | 12 (41.4) | 12 (40) | – | 12 (40) | – | 12 (40) | – |
| III | 9 (31) | 9 (31) | – | 8 (27.6) | 1 (3.4) | 8 (27.6) | 1 (3.4) |
| IV | 5 (17.2) | 4 (13.8) | 1 (3.4) | 5 (16.7) | 1 (3.4) | 5 (16.7) | 1 (3.4) |
| No clasificados | 1 (3.3) | 1 (3.3) | – | 1 (3.3) | – | 1 (3.3) | – |
| Tipo histológico | |||||||
| Adenocarcinoma tipo intestinal | 14 (48.3) | 13 (44.8) | 1 (3.4) | 12 (41.4) | 2 (6.9) | 13 (44.8) | 1 (3.4) |
| Adenocarcinoma tipo difuso | 12 (41.4) | 12 (41.4) | – | 12 (41.4) | – | 12 (41.4) | – |
| Adenocarcinoma tipo mixto | 3 (10.35) | 3 (10.35) | – | 3 (10.35) | – | 3 (10.35) | – |
| Localización del tumor | |||||||
| Fondo | 3 (10.35) | 3 (10.35) | – | 3 (10.35) | – | 2 (6.9) | 1 (3.4) |
| Cuerpo | 9 (31) | 9 (31) | – | 9 (31) | – | 9 (31) | – |
| Antro | 17 (58.6) | 16 (55.2) | 1 (3.4) | 15 (51.7) | 2 (6.9) | 17 (58.6) | – |
| Metástasis | |||||||
| Sí | 6 (20.7) | 5 (16.7) | 1 (3.4) | 5 (16.7) | 1 (3.4) | 6 (20.7) | – |
| No | 22 (75.9) | 22 (75.9) | – | 21 (72.4) | 1 (3.4) | 21 (72.4) | 1 (3.4) |
| No determinada | 1 (3.4) | 1 (3.4) | – | 1 (3.4) | – | 1 (3.3) | – |
CE: cáncer de estómago.
En cuanto a los casos de CCR, se observó que cerca de la mitad tenían un estado avanzado del cáncer, la mayoría correspondían al estadio iv. De los 13 casos con mutaciones en los 3 genes, un alto porcentaje, 77% (10/13), fueron identificadas en adenocarcinomas bien diferenciados. En estos casos, el estadio iv fue el más común (7/13). Las mutaciones se localizaron con mayor frecuencia en el colon descendente (9/13), seguido del ascendente (3/13) (tabla 4).
Descripción general de las características clínico-patológicas y de las frecuencias de mutaciones en los 30 pacientes con CCR esporádico analizados
| n (%) | APC | KRAS | TP53 | ||||
| Normal, n (%) | Mutado, n (%) | Normal, n (%) | Mutado, n (%) | Normal, n (%) | Mutado, n (%) | ||
| Número de pacientes | 30 | 23 (76.7) | 7 (23.3) | 26 (86.7) | 4 (13.3) | 28 (93.3) | 2 (6.6) |
| Edad, años (media) | 62 | 56 | 56 | 63 | 59 | 62.5 | 53 |
| Género | |||||||
| Femenino | 19 (63.3) | 12 (40) | 7 (23.3) | 15 (50) | 4 (13.3) | 17 (56.7) | 2 (6.6) |
| Masculino | 11 (36.7) | 11 (36.7) | – | 11 (36.7) | – | 11 (36.7) | – |
| Estadio del CCR | |||||||
| I | 3 (10) | 2 (6.6) | 1 (3.3) | 3 (10) | – | 3 (10) | – |
| II | 10 (33.3) | 8 (26.7) | 2 (6.6) | 8 (26.7) | 2 (6.6) | 10 (33.3) | – |
| III | 2 (6.6) | 2 (6.6) | – | 2 (6.6) | – | 2 (6.6) | – |
| IV | 10 (33.3) | 6 (20) | 4 (13.3) | 9 (30) | 1 (3.3) | 8 (26.7) | 2 (6.6) |
| No clasificados | 5 (16.7) | 5 (16.7) | – | 4 (13.3) | 1 (3.3) | 5 (16.7) | – |
| Tipo histológico | |||||||
| Adenocarcinoma bien diferenciado | 17 (56.7) | 12 (40) | 5 (16.7) | 13 (43.3) | 4 (13.3) | 16 (53.3) | 1 (3.3) |
| Adenocarcinoma moderadamente diferenciado | 7 (23.3) | 6 (20) | 1 (3.3) | 7 (23.3) | – | 6 (20) | 1 (3.3) |
| Adenocarcinoma mucinoso | 2 (6.6) | 2 (6.6) | – | 2 (6.6) | – | 2 (6.6) | – |
| Carcinoma neuroendocrino | 2 (6.6) | 1 (3.3) | 1 (3.3) | 2 (6.6) | – | 2 (6.6) | – |
| Otro | 2 (6.6) | 2 (6.6) | – | 2 (6.6) | – | 2 (6.6) | – |
| Localización del tumor | |||||||
| Ascendente | 6 (20) | 4 (13.3) | 2 (6.6) | 5 (16.7) | 1 (3.3) | 6 (20) | – |
| Transverso | 3 (10) | 3 (10) | – | 2 (6.6) | 1 (3.3) | 3 (10) | – |
| Descendente | 16 (53.3) | 11 (36.7) | 5 (16.7) | 14 (46.7) | 2 (6.6) | 14 (46.7) | 2 (6.6) |
| Recto | 4 (13.3) | 4 (13.3) | – | 4 (13.3) | – | 4 (13.3) | – |
| Sin clasificar | 1 (3.3) | 1 (3.3) | – | 1 (3.3) | – | 1 (3.3) | – |
| Metástasis | |||||||
| Sí | 12 (40) | 8 (26.7) | 4 (13.3) | 11 (36.7) | 1 (3.3) | 10 (33.3) | 2 (6.6) |
| No | 15 (50) | 12 (40) | 3 (10) | 12 (40) | 3 (10) | 15 (50) | – |
| No determinada | 2 (6.6) | – | 2 (6.6) | – | 2 (6.6) | 2 (6.6) | – |
CCE: cáncer colorrectal.
En resumen, los resultados mostraron una frecuencia total de mutaciones en los genes APC, KRAS y TP53 mayor en el CCR (46.7%) que en la obtenida en las muestras de CE (13.3%) (p=0.006). En las 59 muestras tumorales analizadas no se identificaron mutaciones simultáneas en los 3 genes; solo 6 tumores (10%) contenían mutaciones en 2 genes, distribuidas así: en las muestras de CCR 3 tumores tenían mutaciones simultáneas en APC-KRAS y 2 tumores en APC-TP53, mientras que solo se encontró un caso de CE con mutaciones en APC-KRAS.
Por último, en este estudio se encontró una alta frecuencia de polimorfismos sin diferencias entre los pacientes con CE y CCR (p=0.108). La frecuencia total de polimorfismos para los genes APC, KRAS y TP53 en las 59 muestras tumorales fue del 74.6, el 35.6 y el 35.6%, respectivamente (tabla 2).
DiscusiónEste es el primer estudio de análisis de mutaciones en los genes APC, KRAS y TP53 que se realiza en 59 pacientes con cáncer de estómago y colorrectal en Latinoamérica. En total, se encontró que 18 (30.5%) de las 59 muestras tenían mutaciones.
En las muestras de CCR se encontró una mayor frecuencia de mutaciones de los 3 genes analizados, comparadas con las muestras de CE. Estos resultados son consistentes con los informados por otros autores11,25, en los que se informan que las mutaciones en los genes APC, KRAS y TP53 se presentan con mayor frecuencia en CCR, de acuerdo con el modelo propuesto por Fearon y Volgestein en la secuencia de adenoma-carcinoma13,21. Por el contrario, nuestros resultados en las muestras de CE mostraron que mutaciones en estos 3 genes fueron poco comunes, similar a lo informado en otros estudios3,5,26,27. De lo anterior, podría sugerirse que el CE y el CCR se desarrollan por vías genéticas diferentes, como son las alteraciones cromosómicas, la inestabilidad microsatelital y la metilación en promotores de diferentes genes y alteraciones en otros genes que están relacionados directamente con un mayor riesgo de CE.
En este estudio, el gen APC fue el más frecuentemente mutado de todos los genes analizados en las muestras de CCR, mientras que en las muestras de CE se encontró una baja frecuencia de mutaciones en este gen; sin embargo, esta diferencia no es estadísticamente significativa; este hallazgo es similar a los reportados por Lee et al., quienes encontraron una frecuencia del 2.5% analizando 237 muestras de CE en pacientes de Corea28, y con los de Horii et al. y Yamashita et al., quienes obtuvieron unas frecuencias del 6.8 y 3.4%, respectivamente3,29. Estos resultados en conjunto indican que mutaciones en el gen APC no son comunes en el CE; así mismo, estos trabajos indican una relación negativa entre mutaciones en APC y el desarrollo del CE, lo que concuerda con otros estudios en los que se informa que mutaciones en APC son raras en neoplasias extracolónicas. Dentro de la carcinogénesis del CE se han descrito diversas vías genéticas, como son la inestabilidad cromosómica (CIN), la inestabilidad microsatelital (MSI, que ocurre entre un 30 y 40%), mutaciones en β-catenina y la metilación de los genes APC, MLH1 y RPRM3,6,14,15,30. Por otro lado, en la carcinogénesis del CE se deben tener en cuenta otros factores no genéticos, como la dieta como el alto consumo de carnes rojas, alimentos ahumados y preservados con sal; la exposición a ciertos agentes medioambientales e infecciones con H. pylori, dado que la población colombiana presenta una alta prevalencia de infección por esta bacteria de hasta un 85%10.
Por otro lado, en las muestras de CCR, se encontró una mayor frecuencia de mutación en APC. Sin embargo, la frecuencia reportada en este estudio (26.7%) es menor comparada con las informadas por otros autores, que reportan mutaciones hasta del 60% cuando se analiza toda la región codificante del gen11,19,31,32. En nuestro estudio, solo se analizó en exón 15 de APC, en el que se encuentra la región MCR, y en la que ocurren con mayor frecuencia las mutaciones. También debe tenerse en cuenta que se han reportado diferencias poblacionales en la frecuencia de mutaciones de APC, posiblemente por la composición étnica, el tamaño de la muestra, las mutaciones por fuera de la región MCR y otras alteraciones genéticas en APC, como grandes deleciones y LOH en la región 5q212,11,33,34.
Por otra parte, en las muestras de CE y CCR analizadas se encontró una baja frecuencia de mutaciones en el gen KRAS. La frecuencia reportada para CE concuerda con las informadas en la literatura, que están en el rango de 0 al 10%35-37. En la población asiática, que tiene una alta incidencia de CE, también se reportan bajas frecuencias de mutación para KRAS35. Por lo anterior, se considera que mutaciones en KRAS son muy raras en el CE. En las muestras de CCR, la frecuencia de mutaciones encontrada fue menor de la esperada (13.3%); en la literatura se informa de una frecuencia de mutaciones desde el 26 hasta el 50%31,38,39. Sin embargo, la frecuencia de mutaciones reportada en este estudio es similar a la encontrada por Akkiprik et al. en 43 muestras de CCR esporádico, la cual fue del 12%18. En el CCR se ha observado una alta frecuencia de mutaciones de KRAS en adenomas colorrectales; además, se ha encontrado que las mutaciones podrían perderse a través de la selección en las células que progresan de adenoma hacia carcinoma31,40.
Con respecto a las mutaciones en TP53, en el CCR y el CE se encontró una baja frecuencia de mutaciones, la cual está en el rango reportado en la literatura41. Sin embargo, en las muestras de CCR la frecuencia reportada en este estudio fue sorprendentemente baja comparada con las informadas en otras poblaciones, las cuales son de hasta un 50%42, cuando analizan toda la región codificante del gen. Nosotros evaluamos solamente los exones 5 al 8, que es la región en la que se ha identificado el mayor porcentaje de mutaciones43. No obstante, no se podría excluir la presencia de mutaciones en los exones no evaluados. Las diferencia en las frecuencias informadas podrían explicarse, también, por la exposición a determinados agentes carcinogénicos que inducen determinadas mutaciones puntuales en TP53 y a otras alteraciones genéticas comunes en este gen, como son las microdeleciones y LOH en el locus 17p.13.13,20,42,44-46.
En este estudio, el análisis genético de las muestras de CCR (25 de colon y 4 de recto) reveló que todas las mutaciones detectadas ocurrieron en el colon y se presentaron con una mayor frecuencia en el colon descendente. Por el contrario, en las muestras de cáncer de recto no se encontraron mutaciones, debido posiblemente al bajo número de tumores analizados.
Por otro lado, en el presente estudio no se encontraron mutaciones simultáneas en los 3 genes analizados en las muestras de CE y CC; este resultado concuerda con otros estudios11,12,47. Lo anterior se considera un evento infrecuente, especialmente en los casos de CCR esporádico, en los cuales se ha estudiado ampliamente estos genes, y en los que se reporta una frecuencia de mutación para los 3 genes menor del 7%11,12,47. En solo 6 (10.2%) casos se encontraron mutaciones concomitantes en 2 genes, siendo la combinación APC y KRAS la más frecuente; estos resultados son similares con los reportados en otros estudios12. Lo anterior, podría indicar que las mutaciones en los genes APC y KRAS ocurran en estadios tempranos del CCR y favorezcan la progresión del cáncer12. En relación con el modelo de la carcinogénesis del CCR esporádico, se sugiere que la acumulación de mutaciones en estos 3 genes no es un prerrequisito para el desarrollo de muchos CCR esporádicos, como lo muestra nuestro estudio y los de otros autores, y que al menos una tercera parte de los CCR no siguen este modelo secuencial11,12,47. Por tal razón, se propone que las mutaciones en los genes APC, KRAS y TP53 ocurren por vías genéticas independientes12,18,19.
Finalmente, de forma contraria a las bajas frecuencias de mutaciones encontradas en los genes APC, KRAS y TP53 en las muestras de CE y CCR, en este estudio encontramos una alta frecuencia de polimorfismos en los 3 genes analizados. De los 7 polimorfismos identificados en las 59 muestras analizadas, el de mayor frecuencia fue el rs41115 en el gen APC. Las altas frecuencias de este polimorfismo encontradas en las muestras de CE (86.2%) y de CCR (60%) podrían ser las más altas reportadas hasta el presente en estos 2 tipos de cáncer. Todos los polimorfismos identificados en este trabajo están reportados en la literatura y en la base de datos de SNP del NCBI24,48-52. La asociación de estos polimorfismos con el riesgo de desarrollar cáncer aún no está bien definida53.
El polimorfismo rs41115 se ha identificado en diversos tumores48,49,54-57; particularmente en familias con poliposis adenomatosa familiar (FAP), se ha observado que este polimorfismo cosegrega con mutaciones germinales en APC, por lo que se indica que los individuos de familias con FAP podrían tener un mayor riesgo de desarrollar CCR50. En primer lugar, es importante conocer las variantes genéticas de nuestra población, las cuales no se conocían. Por otra parte, se ha informado la asociación de estas variantes con el riesgo de desarrollar cáncer. Esta población es considerada históricamente como un aislado genético compuesto por una mezcla étnica de europeos, que constituye la mayor proporción, seguida de africanos y en menor proporción de amerindios58; por tanto, estas variantes genéticas harían parte de la composición genética propia de esta población. Son necesarios futuros estudios para validar estos resultados con un mayor número de muestras, como también para esclarecer la asociación de los polimorfismos identificados con el riesgo de desarrollar CE y CCR.
En resumen, este es el primer estudio de análisis de mutaciones en los genes APC, KRAS y TP53 en pacientes con CE y CCR realizado en Latinoamérica. Nuestros resultados aportan información importante para el componente genético de la población colombiana, del patrón de mutaciones y polimorfismos en estos 3 genes. Los resultados de este estudio confirman datos previos de las frecuencias de mutaciones reportadas en CE, pero difieren notablemente con las encontradas en CCR, especialmente con las del TP53. Por último, en la carcinogénesis del CE y del CCR se describen diferentes vías genéticas, como son la MSI, la CIN y la vía epigenética3,6,14,15,30.
ConclusiónLas frecuencias de mutaciones de los genes APC, KRAS y TP53 fueron mayores en las muestras de CCR que en las de CE; nuestros resultados indican la existencia de diferentes vías genéticas en la carcinogénesis del CE y del CCR, y revelan un patrón de mutaciones y polimorfismos particular en los pacientes colombianos estudiados.
FinanciaciónEste trabajo de investigación fue financiado por la Universidad de Antioquia, Medellín, Colombia. Programa de sostenibilidad de grupos 2009-2011 Genética Médica, CPT-0917.
Conflicto de interesesLos autores no tienen conflicto de interés en relación con el artículo que se remite para publicación.
A los pacientes, por su voluntaria participación en este trabajo, y al Hospital Universitario San Vicente Fundación y la Clínica León XIII de la ciudad de Medellín, Colombia; instituciones en las que se recolectaron las muestras de este estudio. Al Dr. Jorge Botero Garcés, por su asesoría en los análisis estadísticos.