El tratamiento para la erradicación de Helicobacter pylori (H. pylori) incluye un inhibidor de la bomba de protones y 2 antibióticos: amoxicilina y claritromicina; este tratamiento tiene como objetivo erradicar la infección en al menos el 90% de los pacientes. El fracaso del tratamiento de erradicación de la infección puede tener múltiples causas, entre ellas la presencia de mutaciones puntuales en los genes diana de los antimicrobianos.
ObjetivoCaracterizar las mutaciones presentes en el gen pbp1a y su posible asociación con la resistencia a la amoxicilina in vitro.
MetodologíaSe evaluó la susceptibilidad a amoxicilina en 147 aislados de H. pylori de Túquerres, se amplificó por PCR y secuenció los dominios transglicosilasa del gen pbp1a en aislados de Túquerres, y se evaluó la asociación entres las mutaciones y la resistencia.
ResultadosSe observó que el 5.4% (8/147) de aislados de Túquerres fueron resistentes a amoxicilina in vitro. La amplificación por PCR de los dominios transglicosilasa del gen pbp1a se realizó en el 87.5% de los aislados resistentes in vitro a amoxicilina, y en el análisis de la secuenciación del ADN se observó un total de 2 cambios de aminoácidos a partir de 3 mutaciones en el ADN que codifica para la proteína PBP1A-1.
ConclusiónEsta investigación constituye el primer reporte sobre las mutaciones en el gen pbp1a en aislados de H. pylori provenientes de las poblaciones de Túquerres; se encontraron mutaciones no reportadas en investigaciones previas.
Helicobacter pylori (H. pylori) eradication treatment includes a proton pump inhibitor and two antibiotics: amoxicillin and clarithromycin. The goal of that treatment is to eradicate the infection in at least 90% of the patients. Failure to eradicate the infection can have multiple causes, among which is the presence of point mutations in the antimicrobial target genes.
ObjectiveTo characterize the mutations present in the pbp1a gene and their possible association with resistance to amoxicillin in vitro.
MethodologySusceptibility to amoxicillin was evaluated in 147 isolates of H. pylori from the Colombian municipality of Túquerres. PCR amplification and sequencing of the glycosyltransferase domain of the pbp1a gene were carried out on Túquerres isolates, and the association between mutations and resistance was evaluated.
ResultsA total of 5.4% (8/147) Túquerres isolates were resistant to amoxicillin in vitro. PCR amplification of the glycosyltransferase domain of the pbp1A gene was performed on 87.5% of the amoxicillin-resistant isolates in vitro, and in the DNA sequencing analysis, a total of 2 changes of amino acids from 3 DNA mutations that encoded the PBP1A-1 protein were observed.
ConclusionThe present study is the first report on pbp1a gene mutations in H. pylori isolates coming from a population in Túquerres. Mutations that have not been reported in previous studies were found.
El Helicobacter pylori (H.pylori) es una bacteria gramnegativa, microaerofílica en forma de espiral, con flagelos que le permiten su movilización y adherencia al epitelio gástrico1. Esta bacteria está asociada a la gastritis y a la enfermedad ulcerosa peptídica, además se considera factor de riesgo en el cáncer gástrico1.
El H.pylori tiene factores de virulencia que le permiten colonizar la mucosa gástrica y desarrollar la enfermedad, que induce a una mayor incidencia y prevalencia de su patogenicidad. Los factores de virulencia más importantes son: la ureasa, el flagelo, el gen cagA, la citotoxina vacA y la proteína babA2.
El H. pylori coloniza la mucosa gástrica de más del 50% de la población del mundo, generalmente se adquiere durante la infancia y persiste durante toda la vida. Cabe resaltar que el resultado clínico de la infección está influenciado por factores ambientales, la diversidad genética de la cepa y la diversidad genética del hospedero3.
En cuanto al tratamiento el esquema de primera línea para la erradicación del H. pylori es la terapia triple, que incluye un inhibidor de la bomba de protones y 2 antibióticos como la amoxicilina y la claritromicina. Este tratamiento tiene como objetivo erradicar la infección en al menos el 90% de los pacientes4; el fracaso del tratamiento de erradicación puede darse por dosis inadecuadas, falta de adherencia al tratamiento, presencia de bombas de eflujo y mutaciones en los genes diana de los antimicrobianos.
La resistencia antimicrobiana es un fenómeno natural que se acelera ante la presión selectiva que genera para los microorganismos la exposición a antibióticos. Lo anterior lleva a que los tratamientos antibióticos convencionales se vuelvan ineficaces, a que las infecciones persistan y a que se incremente el riesgo de su propagación5.
Uno de los antibióticos más ampliamente empleados en la terapia de erradicación es la amoxicilina; este derivado semisintético de la ampicilina inhibe la biosíntesis de la pared celular, uniéndose a las proteínas de unión a la penicilina (penicillin-binding proteins [PBP])6, en la membrana plasmática; PBP1A tiene actividad transglicosilasa/transpeptidasa y su función consiste en el alargamiento de las hebras de peptidoglicano y la formación del entrecruzamiento entre los péptidos de peptidoglucano, por lo que su inhibición interfiere con la síntesis de peptidoglicano y crecimiento celular7.
La prevalencia de la resistencia de H. pylori a la amoxicilina presenta variación geográfica, con valores de 2.2%, 65.6%, 11.6% y de 0.5% para América, África, Asia y Europa, respectivamente8,9. Si bien la información sobre la prevalencia de la resistencia en Colombia es limitada presenta valores del 7% en Bogotá10, del 3.8% en el eje cafetero11 y del 20.5% en Tumaco12; en ninguno de estos estudios se evaluaron mutaciones en el gen pbp1a y su asociación con la resistencia a la amoxicilina. Se desconocen las mutaciones en el gen pbp1a y su asociación con la resistencia a la amoxicilina en aislados de H. pylori provenientes de la población de Túquerres, ubicada en los Andes de Colombia, con prevalencia de H. pylori (>80%) y alto riesgo para el desarrollo de cáncer gástrico13, al igual que la posible asociación entre las mutaciones en este gen y el fracaso en la erradicación de H. pylori. Esta información es útil para el desarrollo e implementación de metodologías de detección rápida de la resistencia y aplicación de esquemas de erradicación, por lo que el objetivo de esta investigación fue caracterizar las mutaciones presentes en el gen pbp1a y su posible asociación con la resistencia a la amoxicilina in vitro.
Materiales y métodosEstudio observacional en el que se incluyeron 149 muestras de mucosa gástrica provenientes de pacientes con síntomas de dispepsia de Túquerres-Nariño, con el propósito de evaluar la resistencia antimicrobiana a amoxicilina in vitro mediante el método de dilución en agar. Para cumplir los objetivos propuestos, primeramente se tomó biopsia de la mucosa gástrica, seguido a ello se realizó cultivo y pruebas bioquímicas para la identificación de colonias puras de H. pylori, lo cual se confirmó mediante la identificación molecular por PCR del gen estructural ureA, seguido a esto se procedió a evaluar la susceptibilidad antimicrobiana a amoxicilina in vitro mediante el método de dilución en agar y, finalmente, a las cepas resistentes se les realizó la amplificación de los dominios transglicosilasa del gen pbp1a y se secuenció para la identificación de mutaciones puntuales. A continuación se detallan los métodos utilizados en este estudio.
Toma de biopsias de mucosa gástricaLa muestra fue seleccionada de adultos de ambos sexos remitidos al servicio médico para evaluación clínica, donde se solicitó una gastroduodenoscopia como parte de su evaluación diagnóstica debido a los síntomas referidos por los pacientes en la consulta clínica, que fueron: dolor abdominal, reflujo gastroesofágico, vómitos, síntomas respiratorios, dispepsia y diarrea. En la tabla 1 se detalla el protocolo de toma de biopsias.
Protocolo de toma de biopsias por endoscopia
| Espécimen | Medio de almacenamiento | Procedimiento |
|---|---|---|
| Dos muestras de mucosas gástrica antral, curvatura mayor y menor | Tiogliocolato con glicerol | Aislamiento de H. pylori y PCR de marcadores de virulencia |
| Cuatro muestras: 2 de antro y 2 de cuerpo gástrico | Formol al 10% | Análisis histopatológico |
| Dos muestras de mucosas gástrica de cuerpo, curvatura mayor y menor | Tiogliocolato con glicerol | Aislamiento de H. pylori y PCR de marcadores de virulencia |
| Una muestra de mucosa gástrica de cuerpo, pared anterior media y otra de antro curvatura menor | Tiogliocolato con glicerol | Preservación a –70°C |
Las muestras se almacenaron y transportaron según las pautas del Ministerio de Salud de Colombia al Laboratorio de Patología de la Universidad del Valle, donde se realizaron los análisis correspondientes.
Cultivo a partir de biopsia gástricaEl cultivo inicial a partir de cual se aisló H. pylori de biopsias gástricas se realizó mediante la introducción de la biopsia en 200μl de NaCl 0.89% estéril, y macerado con macerador estéril; del macerado resultante se tomó con asa de argolla desechable (Fisherbrand® calibrada a 10μl/gota) y se sembró por estría, en agar Columbia (Oxoid®) con sangre desfibrinada de cordero, más 10% suplemento Dent (Oxoid®), el cual contiene vancomicina, cefsulodin, lactato de trimetoprima y anfotericina B. Luego de la siembra se preservó el macerado de mucosa gástrica en una solución de tioglicolato (Merck®) con glicerol (Promega®) 20% y se almacenó a –70°C.
La incubación de las cajas de Petri se hizo en una incubadora de CO2 (Shel Lab®) a 37°C con atmosfera de CO2 al 10% y 90% de humedad. El crecimiento se evaluó a las 72horas y luego periódicamente de 7 a 10 días, en busca de colonias traslúcidas, pequeñas y en forma de gotas de rocío, compatibles con la morfología del H. pylori. Se evaluó la calidad del crecimiento y la contaminación por hongos o bacterias, tanto en el aislamiento primario como en el antibiograma.
Siembra del preservadoPara evaluar la susceptibilidad a amoxicilina en los 147 preservados viables de H. pylori de la población de Túquerres se necesitó aislar nuevamente la bacteria, a partir de sus preservados. Para este fin se tomó una fracción del preservado (–70°C), con lanceta estéril, esta se depositó en agar Columbia (Oxoid®) con sangre desfibrinada de cordero al 10%, sin suplemento. Se sembró en masa e incubó a 37°C en ambiente microaerofílico (CO2 al 10% y 90% de humedad). El crecimiento se evaluó a las 72horas y luego periódicamente de 7 a 10 días. El contenido restante del preservado se almacenó a –70°C.
Purificación de los aislados de Helicobacter pyloriLas colonias obtenidas previamente y que presentaron fenotipo compatible con H. pylori se sembraron por triplicado empleando el método estría con asa estéril (10μl), en agar Columbia (Oxoid®) más sangre desfibrinada de cordero al 10%, con suplemento Dent (Oxoid®), el cual contenía vancomicina, cefsulodina, lactato de trimetoprima, amoxicilina y anfotericina B. Las cajas se incubaron (incubadora Shel Lab®) a 37°C en ambiente microaerofílico (CO2 al 10% y 90% de humedad). El crecimiento se evaluó inicialmente a las 48 a 72h, seguidos de evaluaciones periódicas cada 24h en un periodo de tiempo de 7 a 10 días; a las colonias resultantes se les realizó las pruebas de identificación fenotípica y amplificación del gen ureA.
Identificación fenotípica del Helicobacter pyloriMacroscópicamente se buscaron colonias pequeñas, puntiformes y traslúcidas, con bordes definidos, a las que se les realizaron las siguientes pruebas de identificación: la prueba de oxidasa, catalasa, test de urea (Host Test) y tinción de Gram. Finalmente, para su correcta identificación se compararon con los resultados expuestos en el Bergey'sManual of Systematic Bacteriology para la identificación de H. pylori. Adicionalmente la identificación molecular se realizó mediante amplificación por PCR del gen estructural ureA de H. pylori.
Evaluación de la susceptibilidad de los aislados de H. pylori a amoxicilina mediante el método de dilución en agarLa susceptibilidad antimicrobiana se determinó empleando la metodología de dilución en agar aplicada a los aislados de H. pylori provenientes de los pacientes y donde el punto de corte para considerar una cepa resistente a amoxicilina se definió con una capacidad inhibitoria mínima (CIM) mayor o igual a 1.0μg/ml (Figueroa et al., 2012).
Esta se realizó a los 147 aislados de H. pylori obtenidos a partir del cultivo en agar Columbia base. Para esto se sembró por triplicado y por agotamiento un inóculo de aproximadamente 6×108UFC/ml (McFarland 2), en agar Mueller Hinton (Merck®) suplementado con 10% de sangre desfibrinada de cordero, a concentraciones dobles (0.25, 0.5, 1, 2 y 4.0μg/ml) de amoxicilina, e incubando (incubadora Shel Lab®) a 37°C en ambiente microaerofílico (CO2 al 10% y 90% de humedad). El crecimiento bacteriano se evaluó tras 72h, se empleó la cepa ATCC 43504 de H. pylori como cepa de control de calidad para monitorizar la precisión de la CIM de H. pylori al usar el método de dilución en agar.
Extracción de ADNPara la extracción de ADN bacteriano se tomó 1ml de preservado (80% tioglicolato y 20% glicerol) de H. pylori a –70°C y depositó en tubo Eppendorf de 1.5ml, que seguidamente se centrifugó (centrífuga refrigerada Forma Scientific®) a 13,000rpm durante 2min, se descartó el sobrenadante y se adicionó 300μl de buffer de lisis (Proteinasa K 100μg/ml, SDS 10%, EDTA 0.5M y Tris-HCl pH 8) y se incubó a 56°C por 18h, seguido de 10min a 72°C. Terminada la incubación se adicionó 120μl de NaCl 5M y centrifugó a 13,000rpm por 5min, seguidamente se trasvasó el sobrenadante y descartó el precipitado. Se adicionó 840μl de etanol absoluto (Mallinckrodt®) y centrifugó a 13,000rpm a 4°C por 20min, se descartó el sobrenadante y adicionó 300μl de etanol al 70%, centrifugando a 13,000rpm por 5min descartando el sobrenadante; se dejó secar por espacio de 2h; se resuspendió en 100μl de Buffer TE y almacenó a –20°C. La cantidad y la pureza del ADN bacteriano se determinó por lectura de densidad óptica a 260/280nm en espectrofotómetro (Gene Quant II® Pharmacia Biotech) de acuerdo con las instrucciones del fabricante.
Amplificación del gen ureALa identificación molecular del H. pylori se realizó mediante la amplificación por PCR del gen ureA. La reacción se realizó en un termociclador (Swift MiniProTM, Esco), adicionando los siguientes reactivos a un tubo de 0.2ml: 1X de buffer de PCR (Buffer green 5X Promega®), 1μM de MgCl2 (Promega®), 0.25mM de dNTPs (Promega®), 50pmol/μl de cada cebador (sentido 3’-AAGACATCACTATCAACG-5’/ anti-sentido 5’-CCCGCTCGCAATGTCTAA-3’), 0.5 unidades de GoTaq DNA polimerasa (Promega®) y 25ng de ADN genómico de H. pylori en un volumen final de 25μl. La amplificación se realizó por una desnaturalización inicial a 95°C/2min, seguida de 35 ciclos (95°C/1min, 54°C/1min y 72°C/1min) y una extensión final a 72°C/15min. Los productos de amplificación fueron obtenidos por electroforesis a 80 voltios por 1h en una cámara horizontal (Spectroline Bi-O-Vision®). Estos se visualizaron mediante fluorescencia en luz UV (260/280nm) en un transiluminador (Spectroline Bi-O-Vision®) en geles de agarosa (Sigma®) al 2% teñida con 1μl de bromuro de etidio (0.5μg/ml). Para determinar el tamaño de los amplificados de todos los genes se empleó un marcador de peso de 100pb (Fermentas®); el tamaño del amplicón correspondió aproximadamente a 167pb (fragmento esperado mediante el análisis in silico y respecto a la distancia de migración del marcador de pares de bases).
Amplificación de los dominios transglicosilasa del gen pbp1a en el Helicobacter pyloriLa amplificación de los dominios transglicosilasa (pbp1a) por PCR se llevó a cabo utilizando un termociclador (Swift MiniProTM, Esco) y los siguientes reactivos se agregaron a un tubo de 0.2ml: buffer de PCR 1X (Buffer green 5X Promega®), MgCl2 1mM (Promega®), DMSO al 10%, dNTPs 0.288mM (Promega®), 50pmol/μl de cada cebador pbp1a-1 [R-GCCATTCTTATCGCTCAAGTT y F-TCTCGTGTGAGCACCATG], 0.5 unidades de GoTaq DNA polimerasa (Promega®) y 25ng de ADN genómico de H. pylori en un volumen final de 50μl. El ciclo térmico de amplificación consistió en una desnaturalización inicial a 95°C/2min, seguida de 35 ciclos (95°C/1min, 54°C/1min, 53°C/1min) para pbp1a-1 (dominio transglicosilasa) y 72°C/1min y una extensión final a 72°C/5min.
Los productos de amplificación por PCR fueron obtenidos por electroforesis a 80voltios por 1h en una cámara horizontal (spectroline bio-o-visión®). Estos se visualizaron mediante fluorescencia en luz UV (260/280nm) en un transiluminador (spectroline bio-o-visión®) en gel de agarosa (sigma®) al 2% teñida con 1μl de bromuro de etidio (0.5μg/ml). Para determinar el tamaño de los amplificados de todos los genes se empleó un marcador de peso de 100pb (Fermentas®); el tamaño amplicon fue de 340pb para pbp1a-1dominio transglicosilasa. Los cebadores se diseñaron empleando la secuencia para el gen pbp1a código GenBank: AE000511.1 en el software Primer3V 0.4.014.
Secuenciación e identificación de mutacionesLos amplificados purificados del gen pbp1a se secuenciaron en ambos sentidos (sentido y antisentido) empleando el analizador genético (ABI 3130 Applied Biosystem®) y la metodología Big Dye Terminator (Applied Biosystem®), conforme a las condiciones estandarizadas en el Laboratorio de Genética Molecular Humana de la Universidad de Valle. Se empleó el software BioEdit v 7.1.11® en la edición, alineamiento y traducción de las secuencias. Los cambios en las secuencias se cotejaron mediante el alineamiento local, con la secuencia referencia para el gen pbp1a de la cepa de H. pylori 26695 código GenBank AE000511.1.15.
Se exportó el alineamiento en formato FASTA y se abrió en bloc de notas; la información se copió y pego en un libro de Excel donde se adicionó la formula lógica (DatoX=Dato Y)*1, la cual presentó valores de 0 para datos diferentes entre las secuencias alineadas y 1 para datos iguales. Para cada muestra se determinaron los cambios. Para las muestras de pbp1a adicionalmente se tradujo el ADN a proteína en el software BioEdit, y se realizó el mismo análisis para determinar los cambios.
Análisis estadísticoSe estimó la frecuencia y proporción de aislados resistentes de la población de Túquerres. El rendimiento de la amplificación y secuenciación de los fragmentos del gen pbp1a se evaluó mediante análisis descriptivos. Se estimó la frecuencia de las mutaciones en los genes; para las variables de mutaciones en gen pbp1a solo se realizaron análisis descriptivos por el bajo número de muestras al comparar la resistencia in vivo con in vitro (entre 0 a 3 datos).
Consideraciones éticasEste estudio fue aprobado por el Comité Institucional de Revisión de Ética Humana (CIREH) de la Facultad de Salud de la Universidad del Valle, regulado por la Resolución 008430 del 4/1993 de octubre, emitida por el Ministerio de Salud de Colombia.
ResultadosDe los 149 participantes el 53.7% (80/149) fueron de sexo femenino y 29 de ellas tuvieron edades entre los 18-34 años, mientras que en los de sexo masculino 25 presentaron edades entre los 18-34 años (tabla 2).
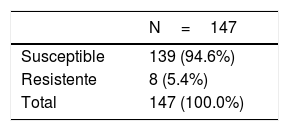
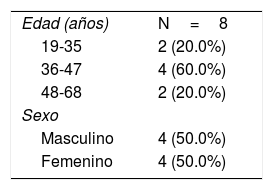
En 147 de los 149 participantes se aisló la bacteria, y al evaluar la resistencia antimicrobiana a amoxicilina in vitro mediante el método de dilución en agar en los aislados de H. pylori se observó que 139/147 (94.6%) de los aislados fueron susceptibles y 8/147 (5.4%) presentaron resistencia in vitro a amoxicilina (tabla 3). La distribución por sexo fue del 50% en los aislados resistentes y fueron obtenidos a partir de pacientes con edades entre 19-35, 36-47 y 48-65 años (tabla 4).
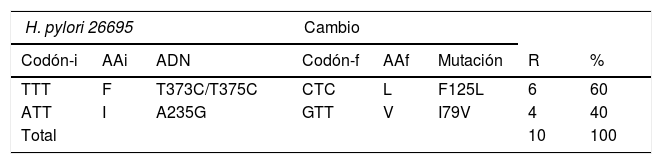
La amplificación por PCR del gen ureA fue positiva para los 147 aislados obtenidos, y la amplificación por PCR de los dominios transglicosilasa (PBP1A) del gen pbp1a de H. pylori se realizó en el 87.5% de los aislados resistentes in vitro a amoxicilina, y en el análisis de la secuenciación del ADN de los 8 aislados resistentes se observaron un total de 2 cambios de aminoácidos. Los cambios en los aminoácidos se presentaron a partir de 3 mutaciones en el ADN que codifica para la proteína PBP1A-1. Los cambios se evaluaron en relación con la secuencia de la cepa de H. pylori 26695, susceptible a la amoxicilina. La mutación F125L se observó en 6 de los aislados, mientras que la mutación I79V se observó en 4 de los aislados (tabla 5).
Descripción de las mutaciones en ADN en las secuencias de pbp1a-1 en los aislados de H. pylori
| H. pylori 26695 | Cambio | ||||||
|---|---|---|---|---|---|---|---|
| Codón-i | AAi | ADN | Codón-f | AAf | Mutación | R | % |
| TTT | F | T373C/T375C | CTC | L | F125L | 6 | 60 |
| ATT | I | A235G | GTT | V | I79V | 4 | 40 |
| Total | 10 | 100 | |||||
AAi: aminoácido original codificado por codón-i; AAf: aminoácido codificado por el codón-f; Codón-f: secuencia del codón mutado en las cepas resistentes de H. pylori; Codón-i: secuencia del codón original en la cepa de H. pylori 26695; R: número de mutaciones observadas en la muestra.
La amoxicilina es un antibiótico comúnmente utilizado en la terapia de erradicación del H. pylori. Aunque la mayoría de los aislados de H. pylori aún son susceptibles a la amoxicilina, la resistencia a este antibiótico está emergiendo en aislamientos clínicos, especialmente en países donde este antibiótico se distribuye ampliamente en la comunidad sin fórmula médica16. Hasta el momento el mecanismo molecular subyacente a la resistencia a la amoxicilina solo se ha identificado para unos pocos aislados de H. pylori resistentes a amoxicilina; en estos aislamientos la resistencia estuvo mediada por mutaciones en el gen que codifica PBP1A16.
Las proteínas de unión a la penicilina o PBP son un grupo de proteínas que se caracterizan por su afinidad y unión a la penicilina, son enzimas que participan en la síntesis y el mantenimiento de la capa de peptidoglicano de la pared celular bacteriana16, y es importante tener en cuenta que hay 2 tipos: PBP1A y PBP2A7. La resistencia se produce a través de modificaciones en las proteínas de unión a la penicilina (PBP), lo que conduce a una disminución de la afinidad por el medicamento. Estas modificaciones incluyen mutaciones y/o mosaicos en PBP2X y PBP2B, así como en PBP1A para los aislamientos altamente resistentes14.
Los estudios orientados a evaluar la susceptibilidad de H. pylori a amoxicilina en Colombia son escasos; estos difieren en la metodología de análisis de la susceptibilidad y en la prevalencia de la resistencia. En esta investigación la prevalencia de la resistencia a amoxicilina en los aislados de H. pylori provenientes de la población de Túquerres fue del 5.4% (8/147), valor muy similar a los reportados en estudios previos; en la ciudad de Bogotá fue reportada la proporción de la resistencia del 7% (6/84) mediante la técnica de difusión en disco10, y del 3.8% (3/79) mediante la técnica de E-test11, y contrasta con lo reportado en aislados de H. pylori del eje cafetero (Armenia y Pereira), donde no se presentó resistencia a la amoxicilina15. Estos registros muestran variación geográfica de la prevalencia en la resistencia a amoxicilina en Colombia, donde los contrastes en la prevalencia de la resistencia pudieran ser explicados por una mayor exposición al antimicrobiano17,18.
En los aislados resistentes provenientes de la población de Túquerres se encontraron variantes y cambios de aminoácidos polares sin carga F125L, I79V. Si bien la asociación de estas mutaciones pudiera estar explicada por su presencia próxima a los sitios de actividad enzimática SKN368-371, SNN433-435, KTG555-557 de la proteína PBP localizados en la región C-terminal, donde las mutaciones provocan cambios conformacionales en las PBP que disminuyen la afinidad de unión a la amoxicilina, dando lugar a fenotipos resistentes19, también pueden asociarse a la presencia de mutaciones como A296V, A494H, A541M, Q572G en gen pbp2, A499V y Q536L en pbp3, los cuales se encuentran fuera de las regiones evaluadas en este estudio20,21.
A pesar de que las mutaciones en el gen pbp1a se asocian con resistencia a la amoxicilina en H. pylori, existen otros genes que pudieran ser responsables del fenotipo resistente, como la presencia de mutaciones en el gen hopC y la deleción en el gen hopB de H. pylori codificante por porinas, que al presentarse permiten a la bacteria sobrevivir a concentraciones de 125mg/l y 250mg/l de amoxicilina respectivamente22. Adicionalmente, si los cambios son simultáneos a mutaciones en el gen pbp1a se observa crecimiento de la bacteria hasta concentraciones de 400mg/l de amoxicilina, lo que sugiere un efecto aditivo23. Entre las características asociadas a la resistencia se encuentra la presencia de betalactamasas, como describe una investigación en la cepa de H. pylori 3778, donde se encontró un producto idéntico a beta-lactamaseTEM-1 (GenBank accession: EU726527) que permitía a esta cepa crecer en medios con concentraciones de amoxicilina mayores 256mg/l24; la resistencia a la amoxicilina es poco probable que se dé por bombas de eflujo, debido a que este antibiótico presenta una hidrofobicidad muy baja, lo que es un requisito indispensable en fármacos que son sustratos de este tipo de estructuras25.
Esta investigación constituye el primer reporte sobre las mutaciones en el gen pbp1a en aislados de H. pylori provenientes de las poblaciones de Túquerres; se encontraron mutaciones no reportadas en investigaciones previas. A pesar de estos resultados se sugiere evaluar el efecto de las mutaciones en diseños de mutación dirigida o modelación, con el fin de identificar su verdadero aporte a la resistencia a amoxicilina.
Fortalezas y limitaciones del estudioEl objetivo de este estudio fue caracterizar las mutaciones presentes en el gen pbp1a y su posible asociación con la resistencia a la amoxicilina in vitro en pacientes de la población de Túquerres, ubicada en los Andes de Colombia, con prevalencia de H. pylori (>80%) y alto riesgo para el desarrollo de cáncer gástrico usando técnicas de identificación microbiológicas y moleculares de H. pylori, que permitieron una mayor seguridad de los resultados obtenidos, aunque se requieren estudios en los cuales se evalúen aislados de las diferentes regiones de Colombia o también de diferentes regiones del mundo, con el fin de identificar mutaciones propias de las cepas circulantes en el medio y su asociación no solo a la resistencia a amoxicilina sino también a otros fármacos empleados en la erradicación para disminuir el fracaso del tratamiento.
FinanciaciónEste proyecto fue financiado por el Departamento Administrativo de Ciencia, Tecnología e Innovación de la República de Colombia-COLCIENCIAS, Beca No. RC: 1902002; y por el Registro Poblacional de Cáncer de Cali, Universidad del Valle, Cali, Colombia.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Al Laboratorio de Microbiología y Biología Molecular, al Laboratorio de Histopatología del Departamento de Patología de la Universidad del Valle que nos permitieron utilizar las instalaciones para desarrollar esta investigación.
Al hospital San José de Túquerres, que nos permitió utilizar sus instalaciones para la toma de las muestras clínicas, de las cuales se obtuvieron los aislamientos de H. pylori empleados en esta investigación.
Agradecimientos a Colciencias y a la Universidad del Valle el apoyo y financiación a esta investigación. Finalmente, al grupo de investigación del Registro Poblacional de Cáncer de Cali y a sus miembros.