La infección por Helicobacter pylori es la enfermedad infecto-contagiosa más diseminada a nivel mundial, alcanzando una prevalencia del 50-80% en países en vías de desarrollo. La infección crónica es considerada como la principal causa de gastritis crónica, y se ha relacionado con otras enfermedades como úlcera péptica, linfoma de tejido linfoide asociado a mucosa gástrica y cáncer gástrico. El tratamiento más común son los esquemas de erradicación que utilizan tres o cuatros fármacos, entre ellos un inhibidor de la bomba de protones (IBP) y dos antibióticos, claritromicina y amoxicilina o metronidazol. El uso empírico de antibióticos para la erradicación de la bacteria ha propiciado una creciente resistencia a dichos fármacos, disminuyendo la eficacia de los esquemas y aumentando los costos para el paciente y para el sector salud. Es por esto que el desarrollo de métodos moleculares no invasivos de siguiente generación promete ser una herramienta que revolucione la terapéutica en H.pylori. La detección genotípica y fenotípica de resistencia a antibióticos de la bacteria permite brindar esquemas personalizados de tratamiento, disminuir costos e implementar un programa de administración de antibióticos.
Los objetivos de esta revisión narrativa son analizar y comparar los métodos diagnósticos tradicionales y de siguiente generación para el diagnóstico de H.pylori, explicar los diversos factores asociados a falla de erradicación y puntualizar sobre el impacto de la creciente resistencia a antibióticos sobre la reversión y la prevención de enfermedades asociados a H.pylori.
Helicobacter pylori infection is the most widespread infectious-contagious disease worldwide, reaching a prevalence of 50-80% in developing countries. Chronic infection is considered the main cause of chronic gastritis and has been related to other diseases, such as peptic ulcer, gastric mucosa-associated lymphoid tissue lymphoma, and gastric cancer. The most common treatment is with eradication regimens that utilize three or four drugs, including a proton pump inhibitor (PPI) and the antibiotics, clarithromycin and amoxycillin or metronidazole. Empiric antibiotic use for eradicating the bacterium has led to a growing resistance to those drugs, reducing regimen efficacy and increasing costs for both the patient and the healthcare sector. In such a context, the development of noninvasive next-generation molecular methods holds the promise of revolutionizing the treatment of H.pylori. The genotypic and phenotypic detection of the resistance of the bacterium to antibiotics enables personalized treatment regimens to be provided, reducing costs and implementing an antibiotic stewardship program.
The aims of the present narrative review were to analyze and compare the traditional and next-generation methods for diagnosing H.pylori, explain the different factors associated with eradication failure, and emphasize the impact of the increasing antibiotic resistance on the reversal and prevention of H.pylori-associated diseases.
Helicobacter pylori es una bacteria gramnegativa espiral y flagelada que coloniza específicamente la mucosa gástrica del ser humano. La infección por H.pylori es la enfermedad infecto-contagiosa más diseminada a nivel mundial; en el año 2015 se estimó que 4.4 mil millones de personas estaban infectadas por esta bacteria, más del 50% de la población mundial, siendo la prevalencia hasta del 80% en los países en vías de desarrollo1. Si bien la tasa de infección es variable, en las últimas tres décadas el número de personas infectadas ha aumentado tanto por el crecimiento poblacional como por falla del tratamiento en su erradicación2; además, se estima que posterior a un tratamiento exitoso la tasa de reinfección es del 2%3.
Es por lo anterior que se decide realizar esta revisión narrativa, cuyos objetivos son analizar y comparar los métodos diagnósticos tradicionales y de siguiente generación para el diagnóstico de H.pylori, explicar los diversos factores asociados a falla de erradicación y puntualizar sobre el impacto de la creciente resistencia a antibióticos sobre la reversión y la prevención de enfermedades asociados a H.pylori. La metodología que se siguió para su realización fue una investigación bibliográfica realizada de forma independiente por dos investigadores en el motor de búsqueda PubMed de ensayos clínicos, revisiones sistemáticas, metaanálisis y guías clínicas publicadas de 1990 a 2021, la cual se centró en métodos moleculares de siguiente generación, resistencia antimicrobiana, nuevos tratamientos y esquemas de administración de antibióticos para H.pylori, utilizando los términos (Helicobacter pylori [MeSH]) AND (next generation sequencing), (Helicobacter pylori [MeSH]) AND (antibiotic* OR antimicrobial*) AND (resistance), (Helicobacter pylori [MeSH]) AND (treatment) y (Helicobacter pylori [MeSH]) AND (treatment) AND (failure).
En cuanto a la prevalencia de H.pylori, existe una gran variabilidad en los diferentes países y regiones del mundo, siendo esta más alta en África (79.1%), América Latina y el Caribe (63.4%) y Asia (54.7%); por otro lado, las regiones con prevalencia más baja son Norteamérica (37.1%) y Oceanía (24.4%)1. Aunque la ruta de infección no ha sido del todo definida, la hipótesis prevalente sugiere una transmisión fecal-oral, donde una madre o abuela infectadas transmiten la bacteria de forma vertical al hijo en los primeros años de vida4. Estas diferencias pueden ser explicadas por los diversos grados de acceso a agua limpia, urbanización, sanidad, estatus socioeconómico, entre otros factores2,5.
En el análisis co-evolucionario de la bacteria y su adaptación en América, un análisis de secuenciación genómica de 723 variedades de H.pylori, apuntó a México como la entrada de la bacteria en el continente6. En cuanto a la población mexicana en particular, la media nacional de seroprevalencia de H.pylori se reporta en el 66%7. Si bien la prevalencia es alta, es notable que se ha encontrado una reducción de la misma, particularmente en niños y en adultos jóvenes. Se cree que esta disminución se debe principalmente a una mejor condición socioeconómica nacional8.
La Organización Mundial de la Salud (OMS) declaró en el 2017 a H.pylori como uno de los 12 «patógenos prioritarios» por su creciente resistencia a antibióticos, y colocó a esta bacteria dentro de la categoría de alta prioridad al ser una amenaza para la salud pública y un carcinógeno claseI para el Centro Internacional de Investigaciones sobre el Cáncer (IARC)9-11. La mayoría de las infecciones cursan asintomáticas —hasta en más del 80% de los casos— y realmente no existe una urgencia para erradicar H.pylori12,13. En caso de ser sintomática, se asocia a enfermedad ulcerosa péptica en el 5-10%, a dispepsia en el 1-10%, a cáncer gástrico no-cardias en <1% y a linfoma de tejido linfoide asociado a las mucosas (MALT) en <0.1%12,14,15. A pesar del bajo porcentaje de pacientes que desarrollan neoplasia, el cáncer gástrico es la tercera causa de muerte relacionado con cáncer a nivel mundial, responsable de 750,000 muertes anualmente, siendo hasta el 90% de los cánceres gástrico no-cardias atribuibles a H.pylori10,16,17. También están bien descritas asociaciones extraintestinales de la infección, como deficiencia de hierro, vitamina B12, púrpura idiopática trombocitopénica, entre otras12,18. Es interesante mencionar que la infección es un factor protector contra el desarrollo de enfermedad por reflujo gastroesofágico, esófago de Barrett y adenocarcinoma esofágico10, y algunos autores incluso cuestionan el rol patogénico de la bacteria, considerándola como parte del microbiota normal de los humanos, siendo un comensal o incluso un simbionte14.
Status quo actual de tratamiento para erradicaciónLas causas de falla en la erradicación de la infección por H.pylori se pueden dividir en factores relacionados al hospedador, relacionados a la bacteria y asociados al sistema de salud.
En cuanto a los factores asociados a la bacteria, la causa más común es la resistencia antimicrobiana. Las tasas de resistencia a antibióticos se estiman, en el caso de la claritromicina, entre el 10 y el 34%; entre el 11 y el 30% para levofloxacino, y entre el 23 y el 56% para metronidazol17. Existe una amplia variación geográfica en cuanto a la tasa de resistencia a antibióticos, y esta ha ido en aumento a lo largo de los años16. En México se reporta una resistencia a claritromicina del 13%, a metronidazol del 60%, a amoxicilina del 4%, a tetraciclinas del 2% y dual a metronidazol/claritromicina del 13%8. Cabe destacar que la resistencia a la amoxicilina y a las tetraciclinas es extremadamente rara, incluso tras la falla de erradicación de la infección posterior a un esquema que incluya estos antibióticos, y la resistencia a bismuto no ha sido descrita a la fecha19. Estas bacterias presentan un fenotipo de resistencia, es decir, pueden crecer ante la presencia del antibiótico y utilizan un mecanismo activo asociado a una mutación heredable para tolerar el estrés inducido por el antibiótico20.
Además del fenotipo de resistencia, existen dos fenotipos más que se asocian a la falla al tratamiento antibiótico: el fenotipo de tolerancia y el de persistencia20. La tolerancia se define como la capacidad de una población bacteriana a sobrevivir a la exposición a concentraciones elevadas de un antibiótico, sin ninguna modificación de la concentración inhibitoria mínima (MIC), por medio del enlentecimiento de los procesos bacterianos esenciales; este fenotipo puede ser adquirido a través de la exposición a condiciones ambientales de estrés21,22. Por su parte, la persistencia es el fenómeno en el que una población bacteriana exhibe características epigenéticas de inactividad y ausencia de actividades metabólicas con el fin de tolerar antibióticos23,24; las características del fenotipo persistente son el cese de la actividad celular, la ausencia de crecimiento o cambio en concentración en presencia del antibiótico y la capacidad de revertir rápidamente al patrón de crecimiento y actividad habitual una vez que se elimina el antibiótico y las condiciones son óptimas para su desarrollo20,24,25. Tanto el fenotipo de tolerancia como el de persistencia son fundamentalmente diferentes al fenotipo de resistencia en que ninguno de estos fenotipos usa un mecanismo activo para tolerar el estrés asociado a un antibiótico, no pueden crecer bajo la presión de dicho medicamento y no se trata de un fenotipo hereditario20. Diferentes mecanismos celulares están implicados en mayor o menor grado en los fenómenos de tolerancia y resistencia; los principales son la respuesta general al estrés, la tolerancia oxidativa, el sistema de comunicación bacteriana (quorum sensing), el sistema de señalización (p)ppGpp y los módulos toxina-antitoxina26. Explicar los mecanismos moleculares detrás de estos procesos está fuera del alcance y de los objetivos de este artículo.
Una característica fenotípica importante de H.pylori es que al activar sus mecanismos de respuesta al estrés puede presentar variabilidad morfológica, es decir, hace una transformación de su forma espiral activa a una forma cocoide inactiva27,28. Las formas cocoides son resistentes a la acidez gástrica y a los antibióticos, permiten la evasión del sistema inmune y se transmiten con mayor eficacia; además, tienen la capacidad de permanecer latentes sin perder su viabilidad ni factores de virulencia, permitiendo una infección crónica y refractaria al tratamiento29,30. Aunado a dificultar el tratamiento, la forma cocoide también representa un reto diagnóstico, ya que no es cultivable, genera falsos negativos en los test de aliento con urea marcada con 13C y pueden causar confusión en la identificación morfológica en biopsias31.
Para lograr una infección persistente, H.pylori cuenta con diversos mecanismos para, a pesar de generar una potente respuesta inmune, evadir, interrumpir y manipular esta respuesta tanto innata como adaptativa del hospedador y así evitar su erradicación32,33. Algunos de los mecanismos utilizados para evadir la respuesta inmune innata incluyen la evasión del reconocimiento y mediación de la señalización por receptores tipo Toll (TLR) y receptores de lectina tipoC (CLR) y el uso de mimetismo molecular34. Para evitar el reconocimiento por TLR, los lipopolisacáridos (LPS) de H.pylori cuentan con cambios estructurales que reducen su detección por estos receptores; estos incluyen su tetra-acilación y la ausencia de grupos fosfato en las posiciones 1’ y 4’ del lípido A35,36. Para modular la respuesta inmune mediada por TLR, la bacteria activa el TLR9, receptor intracelular encontrado en células dendríticas, y el TLR2; ambos desencadenan una respuesta antiinflamatoria37. De los receptores CLR, uno de los mejores descritos es el receptor de lectinaC tipoII, denominado DC-SIGN (Dendritic Cell-Specific Intercellular Adhesion Molecule-Grabbing Nonintegrin, CD-209), encontrado en macrófagos y células dendríticas; los ligandos para este receptor de H.pylori cuentan con residuos de fucosa, y estos residuos activan una respuesta antiinflamatoria32,38. Los antígenos O de los LPS de H.pylori están modificados por fucosiltransferasas que generan estructuras que hacen mimetismo molecular con los antígenos Lewis, evitando la detección por TLR al no ser reconocidos como ajenos39,40. La evasión de la respuesta inmune adaptativa es lograda a través de los factores de virulencia VacA, γ-glutamil transpeptidasa (GGT) y CagA, quienes, a través de diferentes mecanismos moleculares, en conjunto inhiben la proliferación de los linfocitosT34,41. Tanto GGT como VacA también están implicados en la diferenciación de las célulasT hacia linfocitosT reguladores (Treg), y esta inducción es dependiente de la edad a la que el huésped contrajo la infección y es mayor cuando esta ocurre en la infancia, generando tolerancia hacia H.pylori26,42. Otro factor de virulencia asociado a la tolerancia a la infección es la proteína inflamatoria de membrana externaA (OipA), un factor de supresión de la maduración de las células dendríticas, el cual ayuda a establecer una infección crónica por programación tolerogénica43.
En lo que respecta a los factores del hospedador, se han asocian tanto factores genéticos como ambientales a falla en la erradicación de la infección por H.pylori4. Los polimorfismos de CYP2C19 son uno de los principales factores del paciente que se han implicado, ya que la inadecuada supresión de la acidez gástrica es una causa directa de falla al tratamiento, y los polimorfismos de CYP2C19 alteran el metabolismo y, por ende, la efectividad de los inhibidores de la bomba de protones (IBP), especialmente los de primera generación (omeprazol, lanzoprazol)17,44. El gen CYP2C19 tiene 21 polimorfismos, tres de los cuales definen la actividad enzimática que caracteriza el fenotipo metabolizador del huésped a los IBP44,45. Los metabolizadores rápidos son el polimorfismo más frecuente, con prevalencia más alta en caucásicos, afroamericanos e hispanos (57-71%) y menos frecuente en asiáticos17. Es notable que los pacientes homocigotos para el alelo CYP2C19*17 han sido identificados como metabolizadores ultrarrápidos, aunque esta variante no es clínicamente diferente a los metabolizadores rápidos45. Si bien no hay evidencia suficiente que apoye el estudio genético de polimorfismos del gen CYP2C19 en la práctica clínica de rutina para guiar la terapia de erradicación de H.pylori, estos polimorfismos sí tienen implicaciones clínicas, ya que los metabolizadores rápidos y ultrarrápidos se beneficiarían de dosis más altas o de mayor frecuencia en la dosificación de los IBP de primera generación o del uso de IBP más recientes y potentes o, en caso de ser accesible, del uso de supresores de ácido no-IBP, como vonoprazán17,44. Otros polimorfismos genéticos implicados son los que afectan el pH intragástrico, incluyendo de manera importante los genes IL-1B y MDR146.
En cuanto a los factores ambientales, el tabaquismo es el que con mayor frecuencia se ha asociado a falla de la erradicación de H.pylori. El tabaquismo aumenta la secreción de ácido gástrico, reduce la secreción de moco y disminuye el flujo sanguíneo gástrico, reduciendo la efectividad de los IBP y de los antibióticos4,17,47. Otros factores ambientales con menor evidencia de asociación incluyen la edad, la diabetes y la obesidad4.
Además de los factores propios del hospedador y de la bacteria, existen factores del sistema de salud que favorecen la falla a la erradicación de la infección por H.pylori17. Un factor de suma importancia es la adherencia al tratamiento por parte del paciente; se desconoce el nivel de adherencia a partir del cual es despreciable el incremento de la tasa de éxito de erradicación en las infecciones por H.pylori, pero los estudios señalan que la adherencia mínima es de entre el 60 y el 90%48,49. Se han recomendado diferentes estrategias para mejorar la adherencia al tratamiento, como son el usar aplicaciones médicas en teléfonos inteligentes o recordatorios vía mensaje de texto17,50. El médico debe ser claro al explicar el razonamiento detrás de la prescripción de los medicamentos, las instrucciones de dosificación, los efectos adversos esperados y recalcar la gran importancia de completar el esquema completo de tratamiento17. Los efectos adversos relacionados a la terapia de erradicación de H.pylori incluyen náusea, vómito, diarrea y alteraciones en el sentido del gusto, y la presencia de ellos puede llevar a descontinuar el tratamiento antes de terminarlo y contribuir a la falta de adherencia y falla en el tratamiento51. Los costos del abordaje diagnóstico y del tratamiento pueden llegar a ser elevados, dificultando su accesibilidad para algunos sectores de la población19,52.
Métodos convencionales y nuevos métodos moleculares de siguiente generación para el diagnóstico de Helicobacter pylori (tabla 1)Prueba rápida de ureasaLa prueba rápida de ureasa (PRU) es una prueba indirecta de la presencia de H.pylori en la mucosa gástrica. Solo detecta infección activa, a diferencia de la serología. La prueba requiere una biopsia gástrica, que se añade a un dispositivo, donde la muestra se une a urea y luego se detectan los productos de la hidrólisis de urea, amonio o dióxido de carbono, dada la presencia de la enzima ureasa en la bacteria53. La velocidad de la reacción de la PRU depende de la carga bacteriana y de la temperatura. Existen falsos negativos, en el contexto de uso de los IBP o de antibióticos. Los falsos positivos son raros y ocurren cuando hay presencia de otros microorganismos productores de ureasa31, los que, con baja probabilidad, se encontrarán presentes en concentración suficiente para producir un resultado positivo. La PRU no se recomienda para comprobar la erradicación de la infección, salvo que haya indicación de endoscopia digestiva.
Pruebas diagnóstica para Helicobacter pylori
| Prueba | Sensibilidad (%) | Especificidad (%) | Uso |
|---|---|---|---|
| Prueba rápida de ureasa | 80-95 | 97-99 | Requiere de biopsia gástrica. Rápido. Puede tener falsos negativos en contexto de uso de IBP, antibióticos, bismuto o hemorragia digestiva. No se recomienda para evaluar la erradicación |
| PCR | 97-100 | 98 | Permite identificar genes específicos de la bacteria y además evaluar la susceptibilidad antibiótica. Algunos consideran que podría ser el estándar de oro |
| Prueba de aire espirado con urea marcada | 96-97 | 93-96 | Muy buenas sensibilidad y especificidad. Se deben suspender IBP 2 semanas previo al examen dado que disminuyen la sensibilidad. Se puede usar pre/post tratamiento |
| Prueba serológica | 55-100 | 58-96.8 | Varía según el kit usado. Solo se puede usar para monitorización de erradicación |
| Antígeno en deposiciones H.pylori | 83 | 87-94 | Existen pruebas que usan inmunoanálisis enzimáticos y otros inmunocromatográficos. Fácil de implementar. Se puede usar pre/post tratamiento |
Para esta prueba se utiliza la ingestión de urea marcada con 13C o 14C. Si H.pylori está presente, la enzima ureasa de la bacteria libera el CO2 marcado con el isótopo, lo que es medido y comparado con un valor basal. La sensibilidad y la especificidad en general son mayores del 90%54. Se deben suspender los IBP previo al examen debido a que disminuye su sensibilidad. La ventaja de la prueba de aire espirado es que no es invasiva, y puede ser usada, además, para evaluar la erradicación de H.pylori, con un gran rendimiento diagnóstico55.
SerologíaLa serología para H.pylori demuestra exposición al microorganismo y tiene sensibilidad y especificidad variables según el kit serológico utilizado. Una limitación de los estudios serológicos es que no detectan infección activa, por lo que no pueden usarse para monitorizar la terapia56. Su uso es de mayor utilidad en estudios poblacionales de prevalencia de infección por H.pylori.
Antígeno en deposiciones de Helicobacter pyloriEn estos métodos existen pruebas que usan la técnica del inmunoanálisis enzimático y otros que son ensayos inmunocromatográficos rápidos. Algunos utilizan anticuerpos monoclonales y otros anticuerpos policlonales específicos para antígenos de H.pylori57. La ventaja de esta prueba es que es fácil de implementar en distintos centros; además, el antígeno en deposiciones ha sido evaluado en el control de erradicación de H.pylori, presentando buen rendimiento diagnóstico. Se debe tener precaución en pacientes con diarrea, lo que podría disminuir la sensibilidad de la prueba por la dilución del antígeno.
Pruebas molecularesLas pruebas moleculares pueden ser útiles en el diagnóstico de infección por H.pylori. La más utilizada es la reacción en cadena de la polimerasa (PCR), la cual permite, además de la detección de bacterias, evaluar genes patógenos y específicos para la resistencia a antimicrobianos. Para el uso de la PCR se utilizan genes conservados en la bacteria, tales como ureA, ureC, 16SrRNA, 23SrRNA y Hsp6058. Otra ventaja de este método es que se puede extraer la muestra de la biopsia utilizada para la PRU. Algunos estudios han mostrado una sensibilidad para este método de diagnóstico de H.pylori hasta del 100% y una especificidad del 98%59, y algunos autores han planteado que podría ser considerado como el estándar de oro para el diagnóstico, al usar partidores específicos para genes conservados de la bacteria.
Resistencia antimicrobiana y sus mecanismos en Helicobacter pyloriA diferencia de otras bacterias que adquieren resistencia a antibióticos a través de plásmidos transmitidos de manera horizontal, H.pylori tiene una transmisión vertical de mutaciones puntuales involucradas en los mecanismos de resistencia, con un aumento progresivo en la misma debido a la presión selectiva ejercida por el uso de antibióticos31. Los principales mecanismos de resistencia incluyen mutaciones en residuos clave en las proteínas a las que se une el antibiótico, regulación en el sistema de transporte o en la permeabilidad de la membrana para reducir la captación del antibiótico, incremento en la actividad en los eliminadores de oxígeno (oxygen scavengers) y modulación de la función de enzimas involucradas en el metabolismo bacteriano de los fármacos52,60,61 (tabla 2).
Métodos de resistencia a antibióticos de Helicobacter pylori
| Fármaco | Mecanismo de acción61 | Tasa de resistencia América62 | Mecanismo de resistencia52 | Mutaciones detectables por NGS52,60 | Otros posibles mecanismos52 |
|---|---|---|---|---|---|
| MacrólidosClaritromicina | Bacteriostático: unión reversible al dominioV de la peptidil-transferasa de 23S ARNr, interfiere con la síntesis de proteínas | 10% | • Mutaciones en gen que codifica 23S ARNr (rrl), codifica 2 nucleótidos ARNr | rrl codones: 2142, 2142, 2143 | • Mutación hp1048 (infB), hp1314 (rpl22)• Regulación de proteínas de membrana reguladas por hierro, ureasa B, factor elongación Tu, proteínas de membrana externa OMP (HopT, HofC, OMP31) |
| FluoroquinolonasLevofloxacino | Bactericida: inhibición de topoisomerasa tipoII (ADN girasa y topoisomerasaIV) | 15% | • Mutaciones en genes ADN girasa (gyrA, gyrB) | gyrA codones: 86, 87, 88, 91, 97, 130gyrB codones: 463, 481, 484 | • Transmisión horizontal de genes de resistencia a fluoroquinolonas |
| NitroimidazolesMetronidazol | Radicales nitro-anion e intermediarios imidazol que causan daño a estructuras subcelulares y ADN | 23% | • Mutaciones en genes que codifican nitrorreductasa (rdxA, frxA, fdxB)• Mutaciónes en furR3I• Cepas deficientes de recA | No descritas | • Incremento de reparación ADN, deficiencia de transporte• Incremento de la expresión TolC (hp0605, hp0971, hp1327, hp1489) |
| Bismuto | Actividad antimicrobiana contra patógenos gastrointestinales | ? | • Desconocido | No descritas | • No descrito |
| TetraciclinasTetraciclina | Bacteriostático: unión a 16S ARNr de la subunidad 30S del ribosoma, inhibe la síntesis de proteínas | 4% | • Mutación en sitio de unión, región hélix 31 de la molécula de 16S ARNr | 16s ARNr codones: 926-928 | • Bombas de eflujo• Mutación en proteínas de membrana externa hopB o hopC |
| PenicilinasAmoxicilina | Bactericida: unión a proteínas de unión a penicilina (PBP), interfiere con la síntesis de pared bacteriana | 10% | • Mutación en gen PBP1A• Ausencia de gen PBP4 (PBPD) | PPBP1 codones: 402-403, 555-567 | • Mutación en proteínas de membrana externa hopB o hopC |
| RifampicinaRifabutina | Bactericida: unión a la subunidad B de la ARN polimerasa dependiente de ADN, inhibe la transcripción | 1-2% | • Mutación en gen subunidad B de la ARN polimerasa dependiente de ADN (rpoB) | rpoB codones: 525, 545, 585, 149, 701, 149 | • Mutación en codón 701 disminuye MIC |
ADN: ácido desoxirribonucleico; ARN: ácido ribonucleico; ARNr: ácido ribonucleico ribosómico; furR3I: proteína Fur; MIC: concentración inhibitoria mínima; NGS: la secuenciación de siguiente generación (next-generation sequencing); OMP: proteínas de membrana externa; PBP: proteínas de unión a penicilina; recA: proteína recombinación ADN bacteriano.
La claritromicina es un macrólido que se une a la región peptidil-transferasa de la molécula ácido ribonucleico ribosómico 23S (ARNr 23S) en la subunidad 50S ribosomal; su unión inhibe la elongación de las proteínas a través de la liberación prematura del peptidil-ARN del sitio aceptor, bloqueando la síntesis de proteínas52. La resistencia a la claritromicina se genera por mutaciones puntuales en el gen rrl, que codifica los nucleótidos 2142 y 2143 del ARNr 23S63. En orden descendente de frecuencia con la que se observan, las tres diferentes mutaciones presentes en H.pylori resistente a claritromicina son A2143G, A2142G y A2142C60. Las mutaciones A2142G y A2142C se asocian a alta resistencia cruzada con todos los macrólidos, mientras que la mutación A2143G se asocia a resistencia alta a eritromicina y resistencia intermedia a clindamicina y estreptogramina64. Existen otros mecanismos menos importantes por los cuales H.pylori puede adquirir resistencia a la claritromicina, como mutaciones en la bomba de eflujo HefABC (hp0605/606/607), así como en los genes hp1048(infB) y hp1314(rpl22)52,60,64.
El levofloxacino es una fluoroquinolona cuyo mecanismo de acción es la inhibición de la ADN girasa (topoisomerasaII) y topoisomerasaIV52. Estas enzimas son codificadas en 4 genes distintos: gyrA y gyrB en el caso de la ADN girasa y parC y parE en el caso de la topoisomerasaIV (no presentes en H.pylori); las mutaciones en estos genes son las responsables de la mayoría de los casos de resistencia a quinolonas en las bacterias en general65. En el caso específico de H.pylori, las mutaciones en el gen gyrA parecen ser la principal causa de resistencia a fluoroquinolonas, habiendo reportes recientes de mutaciones en el gen gyrB60. Las mutaciones puntuales del gen gyrA en las posiciones que codifican los aminoácidos 86, 87, 88, 91, 97 o 130 son las responsables de la resistencia a las fluoroquinolonas en H.pylori, siendo las más frecuentes las encontradas en los codones 87 y 9152,66,67. En general la resistencia es cruzada con los demás antibióticos de esta familia52.
El metronidazol es un profármaco que debe penetrar a la célula bacteriana para ser activado. Una vez dentro de ella, flavodoxina o ferredoxina oxidados con electrones por el complejo piruvato oxidorreductasa reducen el grupo nitro del metronidazol a radicales aniónicos que inhiben la síntesis de ácidos nucleicos52,60,63,66. El ambiente intracelular de H.pylori es microaerofílico, el oxígeno molecular disponible compite con el metronidazol por electrones, lo cual favorece la formación de superóxidos que resultan en daño al ácido desoxirribonucleico (ADN) de la bacteria65. La resistencia a metronidazol se puede desarrollar por diferentes mecanismos, ya sea por un incremento en la actividad de los eliminadores de oxígeno (oxygen scavengers), por una reducción en la actividad de las nitrorreductasas, una disminución en la captación del antibiótico o un aumento en la actividad enzimática52,60. El mecanismo de resistencia predominante es la reducción de la actividad de las nitrorreductasas, y por consecuencia en la activación del metronidazol. Las mutaciones que inactivan oxidorreductasas como RdxA (nitrorreductasa de NADPH insensible a oxígeno), FrxA (NADP-flavin oxidorreductasa) y FdxB (enzima ferredoxin-like) generan directamente una reducción en la cantidad de enzima nitrorreductasa presente en H.pylori60,63,66. Es importante mencionar que el metronidazol es uno de los pocos fármacos cuya resistencia puede ser afrontada, pues la resistencia in vitro no se correlaciona con la efectividad in vivo, especialmente cuando se usa como componente de terapia triple o cuádruple68.
La amoxicilina es un fármaco del grupo de los betalactámicos cuyo mecanismo de acción es la unión con las proteínas fijadoras de penicilina (PBP) para inhibir la síntesis de la pared bacteriana52,60. En el contexto de su uso para tratar infecciones por H.pylori, es importante destacar que este antibiótico tiene actividad antimicrobiana dependiente de pH, y su MIC es variable en relación al pH en el que se encuentra69. Si bien se ha detectado la presencia de genes β-lactamase-like en el genoma de H.pylori, no se ha encontrado actividad significativa de β-lactamasas en las cepas resistentes a amoxicilina52,60,61. El más importante mecanismo de resistencia a este antibiótico es a través de mutaciones puntuales en los genes que codifican las PBP, principalmente mutaciones en el gen pbp1A que conllevan a sustituciones de aminoácidos en la región transpeptidasa de PBP161. La ausencia del gen que codifica para PBP4 también se ha asociado a resistencia a la amoxicilina63,64,70. Un mecanismo secundario por el cual algunas cepas de H.pylori tienen un MIC aumentado a la amoxicilina es por reducción en la permeabilidad de su membrana a amoxicilina60. Mutaciones en los genes hopB y hopC, que codifican proteínas porinas, causan por sí mismas resistencia a amoxicilina y tienen un efecto sinérgico con mutaciones en PBP65,71.
Las tetraciclinas son antibióticos bacteriostáticos que se unen a la subunidad 30S del ribosoma, inhibiendo la síntesis de proteínas52,60. El principal mecanismo de resistencia a este grupo de antibióticos es a través de mutaciones en la región hélice31 de la molécula ácido ribonucleico ribosómico 16S (ARNr 16S), específicamente en las posiciones 926 a 928, el sitio de unión de las tetraciclinas64,72. Un segundo mecanismo de resistencia descrito en las cepas resistentes a tetraciclinas es la presencia de bombas de eflujo, las cuales disminuyen las concentraciones intracelulares de tetraciclinas52,63.
La rifabutina inhibe la transcripción del ácido ribonucleico (ARN) al unirse a la subunidadβ de la polimerasa de ARN dependiente de ADN RpoB, codificada por el gen rpoB73. La resistencia a este fármaco se debe a mutaciones puntuales del gen rpoB en cuatro diferentes regiones, ya sea en el codón 525545, 585, 149 o 701, siendo las mutaciones en los dos primeros codones mencionados las más comúnmente encontradas52,61,65,73. Las mutaciones se encuentran confinadas en áreas cercanas al sitio activo de RpoB, en donde se une el antibiótico, impidiendo la interacción de la enzima con el fármaco52,73.
Dos mecanismos asociados a H.pylori multidrogorresistente son la formación de biopelículas y las bombas de eflujo74,75. Las bombas de la familia RND tienen particular relevancia, ya que se asocian a resistencia a diversas clases de medicamentos, H.pylori tiene un grupo de 4 genes considerados como bombas de esta familia, estos son Hp0605-0607 (hefABC), Hp0969-0971 (hefDEF), Hp1327-1329 (hefGHI) y Hp1487-1489; el YahR homologo TetA (HP1165) también ha sido implicado60,75. En cuanto a la formación de biopelículas, estas son comunidades de bacterias encapsuladas en una matriz multidimensional de sustancias poliméricas extracelulares que se ha ligado a infecciones crónicas y una reducida susceptibilidad a múltiples clases de antibióticos28,74. Ambos mecanismos generan un fenotipo de resistencia inespecífico para una familia de fármacos, y se ha encontrado que son sinérgicos, ya que las biopelículas de H.pylori tienen una expresión aumentada de bombas de eflujo74.
Nuevas opciones terapéuticas y perspectivas a futuroActualmente los nuevos tratamientos contra H.pylori se basan en metaanálisis de estudios retrospectivos de fármacos en poblaciones seleccionadas hasta conseguir un esquema con mayor tasa de erradicación, sin importar cualquiera que esta sea. Los regímenes que sean porcentualmente superiores, de acuerdo a la guía, se consideran el nuevo status quo de tratamiento. Esto ha impedido por años el desarrollo de nuevas terapéuticas, pues históricamente el desarrollo de antibióticos no es redituable para las farmacéuticas, dado que conlleva años de investigación, son esquemas cortos (7-14días) y se corre el riesgo de que rápidamente se desarrolle resistencia, inhabilitando al fármaco como opción. Debido a lo anterior, nuevas terapéuticas se encuentran en investigación, entre ellas el desarrollo de nuevos fármacos, vacunas, uso de probióticos y nanotecnología.
Dentro de las nuevas estrategias para combatir la resistencia se ha intentado optimizar los factores que influyen tanto en la bacteria como en el hospedador para obtener mejores resultados. La eficacia de los antibióticos radica en su acción en un ambiente gástrico neutro a través de la supresión del ácido gástrico. En este aspecto, se ha referenciado el uso de IBP de segunda generación en dosis altas, pues evitan el metabolismo hepático por CYP2C19 (p.ej., rabeprazol o pantoprazol) y poseen una vida media más larga, aunque tardan el mismo número de días (3-5días) para alcanzar su efecto máximo en el estómago45. Es interesante mencionar que una molécula con un mecanismo novedoso —vonoprazán—, un inhibidor selectivo de canales de potasio aprobado en 2015 en Japón, es hasta 300 veces más selectivo que los IBP tradicionales, alcanza un efecto terapéutico en 1-2días y no se afecta ni se ve influenciado por el metabolismo hepático del huésped45. Esta molécula ha sido aprobada por la Sociedad Japonesa de Gastroenterología, y varios metaanálisis proponen el uso combinado de amoxicilina-vonoprazán como terapia de primera o de segunda línea para infecciones por H.pylori9. Por otra parte, se han desarrollado nuevos antibióticos que todavía no son integrados en los esquemas de terapia triple o cuádruple para H.pylori, entre ellos la sitafloxacina, una fluoroquinolona de cuarta generación, que mantiene actividad bactericida con MIC más altos de resistencia, incluso cuando fallan las quinolonas tradicionales de tercera generación69.
Dado que se usan antibióticos para combatir a la bacteria, surge la hipótesis que la alteración de la microbiota intestinal puede generar una disbiosis que influya a corto y/o a largo plazo en los resultados terapéuticos y pronóstico de los pacientes3. En el corto plazo se ha intentado la suplementación con probióticos, los cuales, al ser bacterias que ofrecen un beneficio al hospedador, pueden influir por sí mismas o a través de la interacción del microambiente en la eliminación de H.pylori51. Sin embargo, un metaanálisis reciente ha demostrado que su suplementación no mejora las tasas de erradicación, aunque pudiera tener impactos benéficos al mitigar los efectos adversos generados por la combinación de medicamentos76. Ahora, en lo que respecta a los cambios de la microbiota a largo plazo, realmente son pocas las especies bacterianas (virales y fúngicas) que logran sobrevivir en el pH gástrico. Un estudio en Lancet, que realizó un seguimiento longitudinal por 1año a pacientes posterior a recibir antibióticos, demostró que las alteraciones de microbiotas presentes a los 14días y 8semanas posterior a iniciar el tratamiento remitieron de forma consistente en el seguimiento a 1año77. Esto indica que los cambios en la microbiota son transitorios, aunque existe evidencia contradictoria de que sí persiste un efecto metabólico (ganancia de peso, síndrome metabólico) a pesar de restaurar la flora intestinal, abriendo la posibilidad de efectos de larga duración posterior al tratamiento15.
En lo que respecta a los nuevos desarrollos tecnológicos, cabe destacar los avances en nanotecnología y los estudios clínicos con vacunas. El uso de la nanotecnología puede proveer nuevas formas de erradicar bacterias al crear compuestos que dañen selectivamente al organismo o alteren las condiciones de su microambiente. El uso de nanopartículas de metales pesados (plata, cobre, zinc) ligadas a una partícula que reconozca selectivamente a H.pylori ha demostrado ser efectivo in vitro al causar lisis del microorganismo por alteración de la pared bacteriana; sin embargo, faltan estudios que avalen su seguridad en humanos78. Asimismo, otra estrategia es acometer contra la formación de biopelículas por H.pylori, pues, en un ambiente estresante, el sistema de comunicación bacteriana (quorum sensing) las protege contra la acción bactericida de los antibióticos, así como induce la transición a su forma cocoide. En este sentido, varias preguntas aún no han sido resueltas en lo que respecta a la nanotecnología74. En lo que concierne a las vacunas, estudios en población pediátrica utilizando los factores de virulencia de H.pylori (p.ej., ompA, cagA) han fallado en mostrar una respuesta duradera y protección a la infección79. Todavía se encuentran en fases primitivas el tratamiento dirigido a contrarrestar factores de virulencia, pero promete ser una nueva revolución terapéutica51.
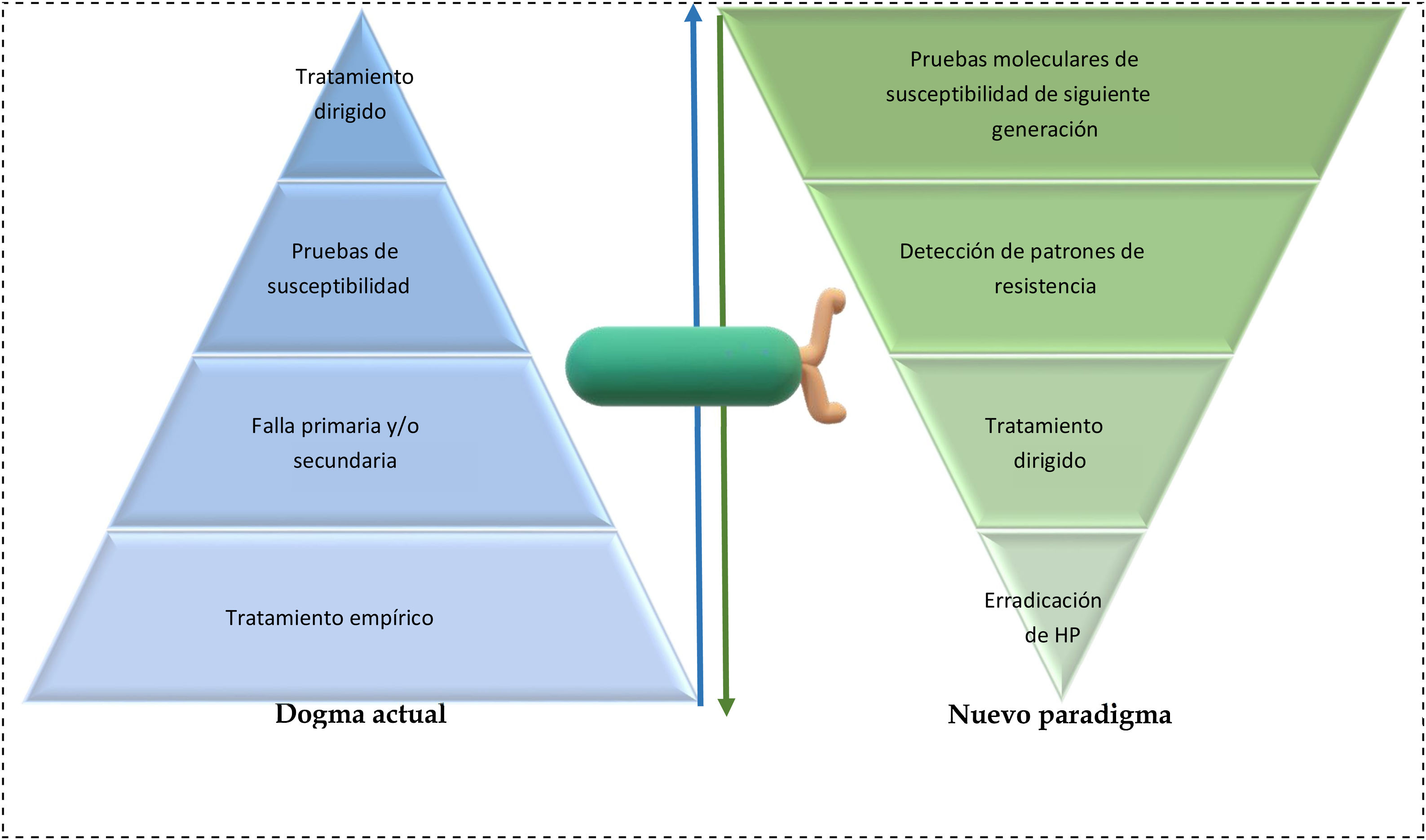
Recapitulando, así como es importante el desarrollo de nuevas terapéuticas, también lo es no perder de vista el horizonte. El problema actual no es la falta de opciones terapéuticas, es la incapacidad para establecer un programa de vigilancia epidemiológica para saber las tasas de resistencia a antibióticos y el uso indiscriminado de esquemas sin eficacia real comprobada. En la actualidad, la mayoría de las guías priorizan la terapéutica con un mayor porcentaje de efectividad y relegan los programas de administración antimicrobiana (antimicrobial stewardship) para cuando todo lo demás falla13,80. Son necesarios más estudios de intención-tratamiento, análisis costo-eficiencia y rentabilidad sobre los métodos moleculares de siguiente generación para lograr un cambio en las guías internacionales. Se tiene que replantear la fórmula, y la secuenciación de siguiente generación (NGS) debe ser el primer paso del algoritmo y no el último (fig. 1).
ConclusionesLa infección por H.pylori se ha convertido en un verdadero reto diagnóstico-terapéutico para los gastroenterólogos. Si bien los métodos diagnósticos tradicionales tienen una sensibilidad y una especificidad aceptables, los métodos diagnósticos moleculares de siguiente generación han demostrado ser altamente sensibles (con una sensibilidad cercana al 100%) y específicos en el diagnóstico de H.pylori, además de brindar el beneficio de identificar genes que confieren resistencia antimicrobiana, permitiendo iniciar un esquema de tratamiento específico para su erradicación, sin comenzar un esquema antimicrobiano empírico al cual la bacteria pudiera ser resistente, como es la práctica actual.
Es indispensable también considerar todos los factores asociados a la falla en la erradicación de la infección, tanto los asociados a la bacteria como los asociados al hospedador, y de manera importante los asociados al sistema de salud. Es de suma importancia para el clínico transmitir al paciente de forma clara y concisa el tratamiento a seguir, advirtiendo de antemano los posibles efectos adversos y poniendo a su disposición herramientas digitales que garanticen la adherencia al tratamiento prescrito.
El aumento en las tasas de resistencia a antibióticos ha generado una creciente falla en las tasas de erradicación de la bacteria, generando un impacto directo sobre la calidad de vida de los pacientes y los costos de atención médica. Los métodos diagnósticos descritos son una estrategia innovadora para el desarrollo de un programa de administración antimicrobiana para la erradicación de H.pylori. Un abordaje de erradicación guiado por susceptibilidad por detección de mutaciones en la sensibilidad a los antibióticos de primera línea es el rumbo a futuro para lograr un cambio en el paradigma de tratamiento de H.pylori.
Consideraciones éticasNo se realizó investigación clínica alguna para la realización de este artículo de revisión, por lo que no fue necesaria la solicitación de consentimientos informados ni de autorización a un comité de ética.
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.
Contribuciones de los autoresGarrido-Treviño, López-Martínez, Tijerina-Rodríguez y Flores-Hinojosa redactaron el artículo y contribuyeron en partes iguales a la elaboración de tablas e imágenes. Bosques-Padilla revisó y elaboró el documento final.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.