La infección por Helicobacter pylori se asocia con gastritis. La variabilidad genética de las cepas tiene importancia clínica. Los estudios de microdiversidad en cepas aisladas de diferente sitio anatómico en un mismo paciente revelan la posibilidad de recuperar 2 o más cepas diferentes.
ObjetivosGenotipificar por PCR multiplex según la virulencia de aislados clínicos de H. pylori para determinar la frecuencia de cada genotipo y relacionarlo con la evolución clínica con la finalidad de prevenir el desarrollo de enfermedades graves.
MétodosSe estudió a 210 pacientes con alteraciones gastroduodenales; 127 cepas fueron identificadas por PCR (glmM y cagE) y después clasificadas según su virulencia por PCR multiplex. Se hizo la prueba de Fisher para evaluar la relación entre genotipo y resultado clínico. La técnica de RAPD-PCR se empleó como método de huella genética y para analizar la presencia de infecciones mixtas.
ResultadoscagA, vacAs1 y vacAm1 estuvieron presentes en todos los aislados clínicos. Las cepas se clasificaron como: tipo i, 40.15%(51/127); tipo ii, 22.04% (28/127), y triples positivas, 28.4% (36/127); se detectaron 2 genotipos nuevos: 1) babA2+, cagA+, vacAs1+, 6.29% (8/27), y 2) babA2+, cagA–, vacAs2/m2+, 3.14% (4/127). cagE se detectó en las cepas tipo ii.
ConclusionesLa prueba de Fisher no mostró una asociación significativa entre el resultado clínico y el genotipo en la población estudiada. Los genotipos circulantes en la población mexicana fueron cagA+, vacAs1, vacAm1. La PCR multiplex puede usarse para genotipificar rápidamente las cepas de H. pylori. cagE es un buen marcador para identificar cepas cag-PAI+.
Helicobacter pylori (H. pylori) is associated with gastroduodenal diseases. Virulence of clinical isolates is related to clinical outcome. Moreover, with microdiversity studies in clinical isolates from a single patient, but from a different origin (antrum or corpus), it is possible to demonstrate that there are simultaneous mixed infections.
AimsTo genotype H. pylori strains with multiplex PCR, according to their clinical virulence, and in this manner know the frequency of each genotype and relate it to clinical outcome in order to prevent the development of severe diseases.
MethodsA total of 210 patients with gastric alterations were studied. Virulence classification of H. pylori strains was carried out with multiplex PCR and 127 strains were identified as H. pylori by PCR (glmM and cagE). Genotype and clinical outcome were evaluated with the Fisher's exact test. In addition, RAPD-PCR was performed as a fingerprinting method to analyze mixed infections.
ResultsThe cagA, vacAs1, and vacAm1 genes were detected in all the clinical isolates. Strains were classified as: type i, 40.15% (51/127); type ii, 22.04% (28/127); and type iii, 28.4% (36/127), but two new different genotypes were also detected: (1) babA2+, cagA+, vacAs1+, 6.29% (8/127) and (2) babA2+, cagA-, vacAs2/m2+, 3.14% (4/127). The cagE gene was detected in type i strains.
ConclusionsThe Fisher's exact test did not support a significant association between clinical outcome and genotype. The main circulating genotypes in the Mexican population studied were: cagA+, vacAs1, and vacAm1. Multiplex PCR can be used as a screening test for H. pylori strains. Furthermore, the cagE gene is a good marker for identifying cag-PAI positive strains.
Helicobacter pylori (H. pylori) es una bacteria gramnegativa, espiriforme, microaerofílica, que se asocia a gastritis y enfermedad ulcerosa péptica (EUP) y se le considera como factor de riesgo en cáncer gástrico1,2. La mitad de la población mundial está infectada con esta bacteria pero solamente el 10% de los infectados presentarán manifestaciones clínicas. La infección por H. pylori puede inducir gastritis crónica, EUP y adenocarcinoma gástrico3. Existe evidencia que muestra la importancia clínica en la variabilidad genética de cepas de H. pylori. Se conocen 3 factores de virulencia como marcadores epidemiológicos y para determinar la presentación clínica: los genes vacA (toxina vacuolizante), cagA (asociado a la citotoxina) y babA (BabA adesina)4.
El gen vacA, que codifica la toxina vacuolizante (VacA), se encuentra en la mayoría de las cepas. Dentro de vacA en la región media (m) se encuentra un polimorfismo significativo con 2 alelos, m1 o m2, así como en la región de la secuencia señal (s) con 3: s1a, s1b, o s2. Los posibles genotipos son: s1bm1, s1am1, s1am2, s1bm2, s2m1 y s2m2, aunque el genotipo s2m1 se reporta como inusual5. El genotipo s1am1, más que el m2, se asocia a EUP3. Una tercera determinante polimórfica de actividad vacuolizante ha sido descrita y se le conoce como la región intermedia (i). Existen 2 subtipos de región i: i1 e i2, y se han observado variaciones entre poblaciones. Los estudios han mostrado una relación significativa entre el alelo i1 y la producción de CagA y la presencia del alelo tipo s1 en varias cepas de H. pylori aisladas en individuos de varios países. Esta asociación podría indicar que la región intermedia tiene un papel importante en manifestaciones clínicas más severas de infección crónica de H. pylori6. CagA se asocia a un riesgo incrementado para cáncer gástrico. CagA es una proteína que es inyectada al interior celular a través de un sistema de secreción tipo IV, en el interior celular es fosforilada e induce cambios morfológicos y probablemente de proliferación celular lo que favorece el desarrollo de gastritis y cáncer gástrico7. Es parte de la isla de patogenicidad cag (cag-PAI) que también contiene el gen cagE, el cual ha sido asociado a EUP en niños y adultos con presentaciones clínicas severas, especialmente en los países desarrollados8,9. BabA media la adherencia de H. pylori al antígeno de grupo sanguíneo humano Lewis-b en células epiteliales gástricas. Tiene un gen silencioso babA1 (no expresado) y un gen babA2 sí expresado. La secuencia de estos genes difiere por una deleción de 10 pares de bases en el péptido señal de babA1. Numerosos estudios indican la relación entre cepas babA2 positivas, con un incremento de mayor densidad de H. pylori e inflamación celular de la mucosa, y un riesgo para desarrollar manifestaciones clínicas severas10-12.
Anteriormente, se clasificaron las cepas como tipo i(cagA+, vacA+) y tipo ii (cagA−, vacA−), de acuerdo con la presencia/ausencia de los genes. Las cepas de tipo i se consideran más virulentas que las cepas tipo ii13. Existe un tercer tipo de cepa, llamada «cepa triple positiva», con la presencia de los genes babA+, vacA+ y cagA+. Se han asociado las cepas «triple positiva» a EUP y adenocarcinoma gástrico14.
Estudios de huella genética han mostrado que cepas independientes de H. pylori se distinguen unas de otras a través de la técnica ADN polimórfico amplificado al azar (RAPD). Los estudios de microdiversidad en aislados clínicos de diferente sitio anatómico en un mismo paciente confirman la posibilidad de recuperar 2 o más cepas diferentes. Mutaciones espontáneas después del pasaje seriado en cerdos gnotobióticos revelaron que H. pylori puede adaptarse al portador permitiendo la transferencia genética intercepas para inducir a la reorganización de su genoma para adaptarse a un nuevo ambiente15-17.
También podrían emerger cepas recombinantes con diferentes combinaciones de genotipos parentales en infecciones mixtas. Varios métodos de huella genética de ADN aplicados a diversos aislados de H. pylori tomados de un mismo paciente muestran que todas las distintas cepas se pueden derivar de una cepa parental ancestral pero que han experimentado cambios independientes en su genoma, un fenómeno conocido como «microevolución». Por lo tanto, es capaz que un mismo paciente sea portador de 2 o más cepas con distinto genotipo17,18.
En México, hay estudios que relacionan la presencia del microorganismo con la manifestación clínica, cáncer gástrico, y con la presencia de infecciones mixtas, obteniendo en un mismo paciente diferentes genotipos19. Un estudio reciente reportó la presencia en la misma cepa de 2 alelos distintos del gen iceA20. El objetivo de nuestro estudio fue genotipificar cepas clínicas de H. pylori a través de PCR multiplex según su clasificación de virulencia y así determinar la frecuencia de cada genotipo y relacionarlo con la presentación clínica con la finalidad de prevenir el desarrollo de enfermedades graves como el cáncer gástrico o la EUP.
MétodosPacientes y muestras clínicas. Se estudió a 210 pacientes con alteraciones gástricas que fueron sometidos a endoscopia gastrointestinal superior en el Hospital General «La Raza» del Instituto Mexicano del Seguro Social en la ciudad de México. Los pacientes de los cuales se aisló H. pylori tenían entre 4 y 17 años de edad. El único paciente adulto del que se aisló H. pylori tenía 62 años. De aquellos pacientes, 11 de 18 eran masculinos y 7 de 18 femeninos. Los diagnósticos predominantes fueron: enfermedad de reflujo gastroesofágico, enfermedad ácido péptica y dolor abdominal recurrente.
Entre enero del 2003 y enero del 2005 se obtuvieron 210 biopsias del cuerpo gástrico de cada uno de los pacientes y fueron evaluadas para la presencia de H. pylori por medio de la prueba rápida de ureasa (PRU) y cultivo. La PRU produjo 30 biopsias positivas (14.3%) del cuerpo gástrico. Por medio de cultivo se recuperó H. pylori en 18 pacientes: 17 fueron niños y un adulto. Previamente al estudio, se obtuvo de cada paciente su consentimiento informado por escrito para la participación en este estudio. Fue aprobado con antelación el protocolo de estudio por el Comité de Ética del Hospital General.
Cultivo de H. pylori y subclonaje. Se cultivó H. pylori de las biopsias. Los especímenes fueron homogenizados en un triturador de tejidos estériles con un diluyente (buffer de fosfato salino, pH 7.5). Los homogeneizados fueron colocados en placas Agar Casman (Difco®) suplementadas con 10% de sangre de oveja desfibrinada y el suplemento selectivo de Skirrow (vancomicina, trimetoprim, polymixina B, OXOID®) y se incubaron a 37°C con un suministro del 5% CO2, el 85%NO2 y el 5% O2 durante 4 días. Colonias típicas fueron identificadas como H. pylori a través de morfología, por coloración de Gram (varillas en forma curva o espiral gramnegativa) y pruebas bioquímicas (ureasa positiva, oxidasa y pruebas de la catalasa). Como se mencionó anteriormente, se cultivó H. pylori ATCC 43504 y se utilizó como el control positivo. Se aislaron un total de 18 cepas clínicas (porcentaje de recuperación, 13.3%) y se les refirió a cada una como un «pool» de cultivo bacteriano. Durante el proceso de aislamiento primario, 4-8 aislados clínicos se tomaron de cada placa por separado y luego se propagaron en placas individuales. A cada una de estas cepas que se obtuvieron se le llamó «colonia individual» o aislado clínico (n=127). Los aislados clínicos se conservaron en criotubos a −70°C en solución suplementada de Hank con 20% glicerol (tabla 1)16,21.
Aislados y subclonas de H. pylori obtenidos de pacientes mexicanos
| Identificación de cepa | Sujeto | Edad (años) | Aislados clínicos | Subclonas RAPD | Genotipo | Diagnóstico |
|---|---|---|---|---|---|---|
| 73 | N | 10 | 8 | 2 | babA2+, cagA+, vacAs1m1+ | GA |
| 126 | N | 14 | 8 | 1 | babA2+, cagA–, vacAs2m2+, | UG |
| B075 | N | 7 | 10 | 3 | babA2–, cagA+, vacAs1m1+, | GN |
| 78 | N | 14 | 8 | 2 | babA2–, cagA+, vacAs1m1+, | UD |
| 94 | N | 11 | 8 | 1 | babA2–, cagA–, vacAs2m2+, | GN |
| 144 | N | 17 | 8 | 1 | babA2–, cagA+, vacAs1m1+, | GN |
| 175 | N | 16 | 6 | 1 | babA2+, cagA+, vacAs1m1+ | GL |
| 39 | N | 11 | 10 | 1 | babA2–, cagA+, vacAs1m1+, | GL |
| B046 | N | 9 | 8 | 2 | babA2+, cagA+, vacAs1m1+, | GL |
| B119 | N | 17 | 7 | 3 | babA2+, cagA–,vacAs1m1+ | UG |
| 58 | N | 8 | 6 | 1 | babA2–,cagA–,vacAs2m2+ | GN |
| 62 | 7 | 4 | 2 | babA2+, cagA+, vacAs1m1+, | GL | |
| 29 | N | 5 | 4 | 2 | babA2+, cagA+, vacAs1+, | GN |
| 36 | N | 17 | 6 | 3 | babA2–,cagA–, vacAs2m2+, | GN |
| 63 | N | 7 | 7 | 1 | babA2+, cagA+, vacAs1m1+ | GN |
| 163 | N | 15 | 7 | 2 | babA2–, cagA+, vacAs1m1+ | GN |
| 110 | N | 5 | 4 | 1 | babA2+, cagA+, vacAs1m1+ | GN |
| 54 | Ad | 60 | 8 | 2 | babA2–, cagA–, vacAs2m2+ | GL |
Ad: adulto; clonas: cepas de subclonaje: GA: gastritis aguda; GL: gastritis leve; GN: gastritis nodular; N: niño o niña; RAPD-PCR: perfil RAPD-PCR diferente; UD: úlcera duodenal; UG: úlcera gástrica.
Extracción de ADN. Fue extraído el ADN de la cepa de referencia y de los aislados clínicos de las colonias y colectado en tubos de microcentrífuga que contenían 100μL de PBS (buffer de fosfato salino, pH 7.5). Las suspensiones fueron agitadas vigorosamente en vórtex por 2min y hervidas en un baño de agua por 15min. Se empleó el método de tiocianato de guanidina para la extracción de ADN. El «pellet» de ADN se suspendió en TE (Tris-EDTA, pH 8.0) y se congeló a −70°C hasta su uso.
Detección del gen glmM. La presencia del gen glmM fue utilizada para monitorizar la pureza de las muestras de ADN y para validar todos los análisis subsecuentes. El ADN genómico de las muestras bacterianas se amplificó con «primers» oligonucleótidos sintéticos GLMM y GLMMR1 (tabla 2), previamente descrito22.
«Primers» utilizados para la amplificación de alelos vacA, cagA, babA2, cagE y glmM por PCR convencional y multiplex
| Gen | «Primer» | «Primer» con secuencia 5′-3′ | Amplicón bp | Referencia |
|---|---|---|---|---|
| vacAm1/m2a | VAGF | CAATCTGTCCAATCAAGCGAG | ||
| VAGR | GCGTCAAATAATTCCAAGG | 570/645 | Atherton et al., 1994 | |
| vacAs1/s2a | VAIF | ATGGAAAATACAACAAACACAC | ||
| VAIR | CTGCTTGAATGCGCCAAAC | 259/286 | Atherton et al., 1994 | |
| cagAa | F1 | GATAACAGGCAAGCTTTTTGAGG | ||
| R1 | CTGCAAAAGAATGTTTGGCAG | 340 | Tummuru et al., 1993 | |
| babA2a | babA2F | AATCCAAAAAGGAGAAAAAGTATGAAA | ||
| babA2R | TGTTAGTGATTTCGGTGTAGGAC | 812 | Mizushima et al., 2001 | |
| cagEb | F1 | GCGATTGTTATTGTGCTTGTAG | ||
| R1 | GAAGTGGTTAAAAATCAATGCCCC | 329 | Tsuneo et al., 2001 | |
| glmMb | GLMMF | GGA TAA GCT TTT AGG GGT GTT AGG GG | ||
| GLMMR | GCA TTC ACA AAC TTA TCC CCA ATC | 140 | Córdova et al., 2011 |
Los perfiles de las huellas genéticas por RAPD-PCR (ADN polimórfico amplificado al azar ADN-PCR). Se llevó a cabo RAPD-PCR para evaluar la variedad genética entre las cepas. La reacción se realizó en 25μL que contenía: 65ng de ADN H. pylori, 0.8μM de los «primers» 1254, 1281, 1283 y 1247, 0.4mM de cada dNTP, 1.5 U de TaqDNA polimerasa (Invitrogen®), y 3mM de MgCl2. El programa de amplificación utilizado fue de 4 ciclos a 94°C, 5min; 36°C, 5min, y 72°C, 5min, y 30 ciclos de 94°C, 1min; 36°C, 1min, y 72°C, 2min, y luego 72°C, 10min en un Perkin-Elmer®, descrito anteriormente22,23. Las bandas RAPD-PCR se evaluaron como presente o ausente y se ensambló una matriz binaria. Las bandas se asignaron según los marcadores moleculares (1 Kb, 100 pb). Se realizó un dendrograma con NTSYSpc-2.02j por el método de grupos de pares con media aritmética no ponderada (UPGMA por su sigla en inglés) y con un coeficiente de asociación sencillo24,25. Finalmente, el coeficiente de correlación cofenético (CCrC) se obtuvo utilizando la prueba de Mantel y la prueba del mejor punto de corte26.
Detección de los genes vacA, cagA y babA2por PCR multiplex. La PCR multiplex se llevó a cabo para genotipificar los genes de virulencia (vacA, cagA, babA2). Fue utilizado el ADN genómico de Proteus mirabilis como el control negativo y H2O como el control en la reacción. Los «primers» utilizados para la PCR se muestran en la tabla 2. Se llevó a cabo amplificación en una reacción con un volumen de 25μL que contenía 10 pM del «primer» babA2 y 5 pM de los «primers» cagA y vacA, 10 X solución Buffer (Invitrogen®), 1.5mM MgCl2, 25mM dNTPs (Invitrogen®), 80ng de plantilla de ADN y 1 U Taq de ADN polimerasa (Invitrogen®). Los ciclos se realizaron en un termociclador Perkin Elmer® con un ciclo inicial de desnaturalización a 94°C por 5min, seguido de 35 ciclos a 94°C por 30 s, 57°C por 45 s y 72°C por 1min. Se distinguieron los amplicones por su tamaño después de la separación por medio de electroforesis en una agarosa de 1.5% y revelados con bromuro de etidio (0.5μg/mL).
Los amplicones H. pylori ATCC 43504 obtenidos por PCR multiplex fueron purificados con un kit Marligen (Bioscience®) en acuerdo con las instrucciones del fabricante y secuenciados en la Unidad de Síntesis y Secuenciación IBT, UNAM, en Cuernavaca, Morelos, para validación PCR multiplex. Las secuencias fueron comparadas con las secuencias reportadas en la literatura médica publicada en la base de datos de la NCBI por análisis BLAST. Adicionalmente, se llevaron a cabo los métodos Southern Blot y Dot Blot para comparar y validar los resultados obtenidos de la PCR.
Detección del gen cagE. Se realizó la amplificación del gen cagE como marcador cag-PAI, en vez de cagA. Los «primers» utilizados se muestran en la tabla 2. Se realizó la amplificación en una reacción con volumen de 25μL que contenía: 10 pM de cada «primer», 10 X solución Buffer (Invitrogen®), 1.5mM MgCl2 (Invitrogen®), 25mM dNTPs (Invitrogen®), 80ng ADN y 1 U Taq de polimerasa de ADN (Invitrogen®). El protocolo de PCR incluyó un ciclo inicial de desnaturalización a 94°C por 5min, seguido de 35 ciclos a 94°C por 60 s, 56°C por 60 s y 72°C por 1min.
Análisis de datos, análisis univariados. Se llevó a cabo una matriz de genotipos para todos los genes codificados como presente (1) o ausente (0). Se calculó el porcentaje de alelos positivos (presente) para cada gen a partir de los datos de todas las cepas. Para evaluar si existía variación entre la PCR convencional y multiplex en la detección de genes de virulencia (babA2, cagA, vacAs1m1), se obtuvo el porcentaje de verdaderos y falsos verdaderos usando la prueba de la chi al cuadrado. Para valorar si la presencia o ausencia de los alelos (vacAs1, vacAs2, vacAm1, vacAm2, cagA, babA2) se asociaba a la presentación clínica (gastritis aguda, úlcera duodenal, úlcera gástrica, gastritis nodular y gastritis leve), se realizó una prueba de Fisher para cada gen usando una tabla de contingencia de 2×2 en relación con el número de cepas, con o sin alelo, y la presentación clínica27.
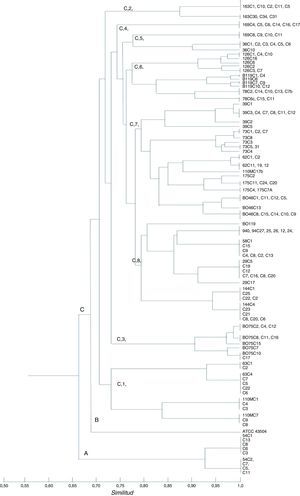
ResultadosRAPD-PCR. El perfil RAPD-PCR reveló 3 a 17 bandas para cada cepa que se analizó. Los clones aislados de cada paciente se mencionan en la tabla 1. La RAPD-PCR detectó la presencia de múltiples cepas de H. pylori, cada una diferente de la otra, en una misma persona. Cuatro «primers» produjeron marcadores genéticos y perfiles RAPD reproducibles (la genotipificación de virulencia se realizó solamente para 127 cepas) entre las cepas estudiadas. El análisis RAPD agrupó cepas basado en la enfermedad clínica. La mayoría de los niños estaban colonizados por una sola cepa. La información sugiere, aunque la población H. pylori consistía de bacterias estrechamente relacionadas, que no era completamente homogénea; variantes genéticas de las mismas cepas de H. pylori pueden existir entre los aislados. Dos poblaciones de H. pylori con distintos patrones RAPD colonizaron a 7 niños. Sin embargo, 3 niños fueron colonizados por clones más estrechamente relacionados con cepas de individuos independientes que entre ellos mismos (tabla 1; figs. 1 y 2). Los resultados del estudio indicaron que hubo una colonización de múltiples cepas en niños mexicanos. El CCrC fue 0.86801 (rango 0.6-0.9), mostrando una representación significativa de la relación genética entre los aislados.
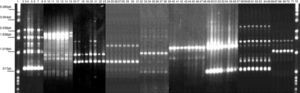
RAPD-PCR, 1281 oligonucleótido. 1Kb escalera, marcador de talla molecular, carril 1, reactivos de control, 2-8: Bx78 (cepas 78C2, 7b, 10, 11, 13, 14); 9-15: 63 cepa (63 C1, 2, 4, 5, 6, 7, 22); 16-23: B046 cepa (B046 C1, 5, 9, 10, 11, 13, 14, 15); 24-31: 29 cepa (29 C5, 7, 8, 12, 16, 17, 19, 20); 32-38: 58 cepa (58 C1, 2, 4, 8, 9, 13, 15); 39-48:144 cepa (144 C1, 2, 4, 6, 8, 20, 21, 22, 23, 25); 49-57: 54 cepa (54 C1, 2, 3, 5, 6, 7, 8, 11, 13); 58-65: 163 cepa (163 C1, 2, 5, 10, 11, 30, 31, 34); 66-71: 175 cepa (175 C2, 4, 7a, 11, 20, 24); 72: escalera marcador de talla molecular 1Kb.
Dendrograma RAPD-PCR de cepas de H. pylori. Tres racimos separados se indican con A, B y C. El racimo A muestra la cepa adulta (54); el racimo B consiste de la cepa ATCC 43504; el racimo C consiste de las cepas pediátricas mexicanas (73, 126, B075, 78, 94, 144, 175, 39, B046, B119, 58, 62, 29, 36, 63, 163, 110 y 169); 169 cepas no se genotipificaron. El grupo C muestra 8 racimos. El CCrC fue 0.86801. Fueron utilizados el programa pasado y el coeficiente de asociación sencillo.
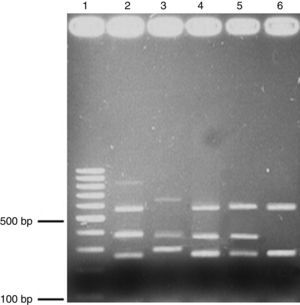
Evaluación por PCR multiplex. Ciento veintisiete cepas clínicas de H. pylori obtenidas de una población pediátrica se caracterizaron por PCR convencional y multiplex (fig. 3) y los resultados de los métodos Southern Blot y Dot Blot fueron correlacionados. La capacidad de detección del Dot Blot fue: cagA (100pg/μl), vacAs1 (100pg/μl), vacAm1 (100pg/μl) y babA2 (1pg/μl). Ambos métodos detectaron el mismo número de positivos verdaderos y/o cepas negativas para vacAs1m1, vacAs2m2 y cagA. Sin embargo, el gen babA2 tuvo una diferencia estadísticamente significativa (chi al cuadrado) (χ2) (tabla 3).
PCR multiplex. Electroforesis en gel de agarosa (1.7%); muestra los amplicones obtenidos a través de PCR multiplex. Carril 1, 100-bp escalera; carril 2, control positivo de cepas de virulencia triple, carriles 3-6, aislados clínicos. babA2 (812bp); vacAs1 (259bp); vaAs2 (286bp); vacAm1 (570bp); vacAm2 (645bp) y cagA (340bp). El ADN genómico de la cepa H. pylori ATCC 43504 se utilizó como control positivo.
Comparación de detección de gen de virulencia entre PCR convencional y multiplex
| Gen/alelo | Valor | Convencional | Multiplex | χ2 | P |
|---|---|---|---|---|---|
| babA2 | P | 48 | 15 | 82.323 | 1.16×10−19 |
| N | 79 | 112 | |||
| cagA | P | 84 | 87 | 0.32845 | 0.56657 |
| N | 43 | 40 | |||
| vacAs1 | P | 91 | 91 | 0 | n.s. |
| N | 36 | 36 | |||
| vacAs2 | P | 36 | 36 | 0 | n.s. |
| N | 91 | 91 | |||
| vacAm1 | P | 87 | 87 | 0 | n.s. |
| N | 40 | 40 | |||
| vacAm2 | P | 36 | 36 | 0 | n.s. |
| N | 91 | 91 |
N: valor negativo; n.s.: no significativo; P: valor positivo; χ2: estadística de chi al cuadrado.
Obtuvimos valores falsos negativos cuando comparamos los resultados por PCR convencional.
Cepas H. pylori: clasificación y genotipificación. Se detectó una alta cantidad de los genes cagA, vacAs1 y vacAm1 en todos los aislados clínicos. Los resultados de los genes se muestran en la tabla 4. Las cepas genotipificadas por PCR multiplex se clasificaron como tipo i, tipo ii y triple positivas, clasificados así por Atherton et al.5 y Gerhard et al.14. Encontramos 2 genotipos diferentes de las cepas del tipo i, ii y triple positivas, que se describieron como «no tipificable». En una cepa (B119) y sus aislados clínicos (n=7), el gen cagA fue negativo utilizando PCR convencional o multiplex, pero detectable cuando se usó el método Dot Blot, por lo tanto, todos se clasificaron como cepas triple positivas. Los genotipos vacAs1 y vacAm1 estuvieron presentes en la mayor parte de las presentaciones clínicas y los genes cagA fueron asociados con mayor frecuencia a gastritis y enfermedad de úlcera gástrica. El gen babA2 se encontró en pacientes con úlcera gástrica y gastritis leve, pero la prueba de Fisher no apoyó una asociación significativa entre la presentación clínica y los genes estudiados (tabla 5). El gen babA2 se detectó en 48 aislados clínicos y se distribuyó principalmente en cepas triple positivas (n=29). El gen babA2 también se detectó en las cepas «no tipificables» mencionadas anteriormente de la siguiente manera: babA2+, cagA+, vacAs1+ (n=8), babA2+, cagA−vacAs2/m2+ (n=4). Cada paciente estaba infectado con solo un tipo de cepa (tipo i, ii, triple positiva o con la cepa «no tipificable») (tabla 6). El gen cagE se amplificó por PCR. La amplificación del gen cagE se ha utilizado para evaluar la presencia o ausencia de cag-PAI, ya que la ausencia del gen cagA en cepas H. pylori no significa que el cag-PAI está del todo ausente. El gen cagE se detectó en todas las cepas de tipo ii de pacientes con enfermedad de úlcera (100%), por lo tanto, el gen cagE puede ser útil y es mejor marcador que el gen cagA en la detección de la presencia de la isla de patogenicidad completa cag-PAI.
Porcentaje y cantidad total de los marcadores de virulencia de cepas pediátricas de H. pylori babA2, cagA y vacAs1m1
| Gen/alelo | Cantidad total | 95% límite de confianza | Porcentaje |
|---|---|---|---|
| vacAm11 | 89 | 61.3-77.9 | 68.5 |
| vacAm2 | 34 | 19.3-35.4 | 28.34 |
| vacAm− | 4 | 0.9-7.9 | 3.16 |
| vacAs1 | 93 | 64.6-80.7 | 71.63 |
| vacAs2 | 34 | 19.3-35.4 | 28.34 |
| cagA+ | 84 | 57.2-7.3 | 66.14 |
| cagA− | 43 | 25.7-42.8 | 33.86 |
| babA2+ | 48 | 29.3-46.8 | 37.79 |
| babA2− | 79 | 53.2-70.7 | 62.21 |
Altos porcentajes. Programa EpiInfo™ 2000.
babA2: gen codificante de BabA adesina dirigido a antígenos del grupo sanguíneo Lewis-b (α-1, 3/4-difucosilados); cagA: gen codificante de una proteína CagA altamente inmunogénica; vacA: gen de citotoxina vacuolizante. En negrita: porcentajes más altos de cada gen o alelo.
Asociación entre virulencia de gen y presentación clínica
| Gen % | GA n=1/18 | UD n=1/18 | UG n=2/18 | GN n=9/18 | GL n=5/18 | Cantidad total |
|---|---|---|---|---|---|---|
| vacAm1 | 1(100)* | 1(100)* | 1(50)* | 5(55.5)* | 4(80)* | 12 |
| vacAm2 | 0(0)* | 0(0)* | 1(50)* | 3(33.3)* | 1(20)* | 5 |
| vacAs1 | 1(100)* | 1(100)* | 1(50)* | 6(66.6)* | 4(80)* | 13 |
| vacAs2 | 0(0)* | 0(0)* | 1(50)* | 3(33.3)* | 1(20)* | 5 |
| cagA | 1(100)* | 1(100)* | 0(0)* | 6(66.6)* | 4(80)* | 12 |
| babA2 | 0(0)* | 0(0)* | 2(100)* | 3(33.3)* | 3(60)* | 8 |
GA: gastritis aguda; GL: gastritis leve; GN: gastritis nodular; UD: úlcera duodenal; UG: úlcera gástrica;
Clasificación de los genotipos de las cepas de H. pylori
| Clasificación | Genotipo | Cepas totales n=127 (%) | RC | Número de pacientes |
|---|---|---|---|---|
| I | babA2−, cagA+, vacAs1m1+ | 51 (40.15) | GA,GN,UD, GL | 6 |
| II | babA2−, cagA−, vacAs2m2+ | 28 (22.04) | GN, GL | 4 |
| III | babA2+, cagA+, vacAs1m1+ | 29 (22.83) | GN, GL | 5 |
| 1 | babA2+, cagA+, vacAs1+ | 8 (6.29) | GN | 1 |
| 2 | babA2+, cagA−, vacAs2m2+ | 4 (3.14) | UG | 1 |
| 3 | babA2+, cagA-,vacAs1m1+ | 7 (5.51) | UG | 1 |
GA: gastritis aguda; GL: gastritis leve; GN: gastritis nodular; RC: resultado clínico; UD: úlcera duodenal; UG: úlcera gástrica.
Existen muchas ventajas en el empleo de la PCR multiplex. Un ejemplo es cuando se trabaja con microorganismos exigentes o nosocomiales, ya que esta metodología incrementa la efectividad de detección y reduce el costo de examinación. La PCR multiplex puede ser útil para genotipificar aislados clínicos obtenidos de biopsias o cultivos y diagnosticar efectivamente la presencia de una infección por H. pylori, pero también para diferenciar las especies de Helicobacter aparte de H. pylori28,29. En nuestro estudio, la sensibilidad y la especificidad de la PCR multiplex fueron excelentes para todos los genes. La PCR multiplex puede utilizarse como un examen discriminatorio para distinguir las cepas tipo i, que son más virulentas y que se asocian a desenlaces clínicos más severos. Algunos autores han descrito que puede existir una relación entre genotipo de la cepa y la presentación clínica, permitiendo la predicción del segundo30. Otros autores han reportado que cepas triple positivas (babA2+, cagA+, vacAs1m1+) se asociaban a úlcera duodenal y adenocarcinoma14. En poblaciones adultas, cepas triple positivas se vinculan a enfermedades severas. Sin embargo, no encontramos relación alguna entre genotipo de la cepa y la presentación clínica en la población pediátrica que se estudió. De acuerdo con nuestros resultados, los aislados clínicos de tipo ii se relacionaban más a gastritis no atrófica.
Los aislados clínicos se clasificaron como tipo i (n=51), tipo ii (n=28) y triple positivas (n=29). Usamos la valoración Dot Blot para detectar el gen cagA en la cepa B119 (n=7) porque no fue posible detectar el gen por PCR convencional ni multiplex. Por lo tanto, la cantidad de cepas triple positivas (n=36) se incrementó. Diecinueve aislados clínicos no se pudieron genotipificar utilizando la clasificación convencional (tipo i, ii o triple positiva), entonces los nombramos cepas «no tipificables»: babA2+, cagA+ y vacAs1+ (n=4) y babA2+, cagA− y vacAs2m2+ (n=8).
La variabilidad del gen vacA se ha asociado previamente con regiones geográficas que incluyen Colombia, la región sureste de Brasil y los Estados Unidos, así como otras poblaciones occidentales, y cepas con un genotipo vacAs1m1 se consideraron productoras más altas de citotoxina, asociadas en lo principal con niños sintomáticos, en pacientes con EUP y en pacientes con dispepsia funcional. Sin embargo, en China, Japón y Corea del Sur no es tan predominante este genotipo y no existe relación31. En el presente estudio, la forma alélica m1 del gen vacA estaba en todos los aislados clínicos y en aquellos pacientes con EUP (68.5%). Los pacientes infectados con cepas que tenían la forma alélica s1 (71.63%), así como cepas conteniendo los genotipos vacAs2m2 y vacAs1m1, se correlacionaron con gastritis aguda, gastritis leve, gastritis nodular y úlcera duodenal. Sin embargo, no se encontró una relación concluyente entre el genotipo de la cepa y la presentación clínica. En nuestro estudio, las cepas cagA+ (66.14%) se aislaron de pacientes con gastritis y enfermedad ulcerosa péptica.
Detectamos cepas tipo ii (n=28) aisladas de pacientes con gastritis nodular y leve. La amplificación del gen cagE se realizó para verificar la presencia de cag-PAI, ya que este gen se encuentra en todas las cepas tipo ii. Dada que la ausencia de cagA no significa necesariamente la falta, la interrupción o la pérdida parcial del cag-PAI, amplificamos el gen cagE, utilizándolo como un marcador opcional para verificar la falta de cag-PAI y finalmente correlacionar las cepas tipo II, cagE+, y desenlaces clínicos severos. En nuestra población, el gen babA se detectó con un porcentaje bajo (37.79%). En contraste, estudios sudamericanos alternos han reportado una frecuencia que va del 46 al 82.3%31. En nuestro estudio, el gen babA se encontró principalmente en pacientes con gastritis (n=6) y EUP (n=2), al igual que ha sido reportado en otros países32,33.
Cepas tipo i (n=51) se aislaron de pacientes con gastritis (n=5) y enfermedad de úlcera duodenal (n=1). Nuestros resultados son similares a aquellos reportados en una población occidental, en la cual el predominio de este genotipo era del 60-90%. Sin embargo, el tamaño de la muestra debe de ser incrementado para así establecer una relación entre el genotipo de cepa y la presentación clínica.
Las cepas triple positivas (cagA, vacAs1m1, babA2) se aislaron de pacientes con gastritis (n=5), pero no pudimos establecer una relación entre el genotipo y la presentación clínica. Aun así, en varios estudios en Colombia fue posible correlacionar la EUP y el carcinoma gástrico con estos genotipos14,31. En países occidentales, estos tipos de cepas son un factor de riesgo, pero no se ha encontrado una relación con desenlaces clínicos severos en poblaciones japonesas. En el presente estudio, todos los aislados clínicos se analizaron por RAPD-PCR y en un mismo paciente observamos algunos aislados clínicos con diferentes patrones de huella genética, indicando que el paciente se presentó con una infección mixta (2 cepas con diferencias genéticas derivaron de la cepa 73). Todos los resultados de la PCR multiplex de estos distintos aislados de un mismo paciente tenían el mismo genotipo (babA2, cagA, vacAs1m1). Se observaron resultados similares en los aislados clínicos 39C5, B119C2, 29C17, 36C10 y 110M17.
La cepa 126 y sus clones respectivos (126C1, 126C2, 126C3, 126C4, 126C6, 126C7, 126C10 y 126C16) fueron aislados de un paciente de 14 años de edad con úlcera gástrica. En esta cepa, se observó el mismo patrón RAPD de huella genética con el mismo genotipo (babA2+cagA−, vacAs2m2+). Por lo tanto, el gen babA+ en estas cepas podría incrementar el factor de riesgo para el desarrollo de úlcera gástrica.
En conclusión, la PCR multiplex se ha utilizado como un rápido ensayo para la detección de más de un factor de virulencia en una sola reacción y también como un método para diagnosticar enfermedades, reduciendo los costos de los exámenes para los pacientes y mejorando el tratamiento. Nuestro estudio propone el uso de la PCR multiplex como una estrategia de cribado para aislados clínicos de H. pylori y clasificarlos de acuerdo con los factores de virulencia de una manera rápida y eficaz, para prevenir el desarrollo de enfermedades severas y para ser utilizadas como una herramienta de monitorización clínica para pacientes que ya han sido diagnosticados. Los resultados obtenidos en este estudio muestran una variedad de genotipos de virulencia en México. El genotipo de cepa prevalente que circula en la población mexicana fue el tipo I (babA2−, cagA+, vacAs1m1) y se encontró en todas las presentaciones clínicas. El gen cagE era un mejor marcador que el cagA en la detección de cepas cag-PAI-positiva.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que los procedimientos seguidos se conformaron a las normas éticas del comité de experimentación humana responsable y de acuerdo con la Asociación Médica Mundial y la Declaración de Helsinki.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores han obtenido el consentimiento informado de los pacientes y/o sujetos referidos en el artículo. Este documento obra en poder del autor de correspondencia.
FinanciamientoSe recibió apoyo financiero para este estudio de las instituciones SIP-IPN (20130644), CONACYT, SNI, EDI, y COFAA.
Conflicto de interesesLos autores declaran no tener algún conflicto de interés.
Los autores agradecen al Instituto Mexicano del Seguro Social y al Dr. Consuelo Ruelas-Vargas del Departamento de Endoscopias Pediátricas del Hospital General «La Raza» por la ayuda recibida en la recolección de especímenes.