En los últimos años, los probióticos se han utilizado en trastornos gastrointestinales funcionales, incluido el estreñimiento crónico (EC). El efecto de la cepa 35624 de Bifidobacterium infantis sobre la microbiota intestinal de pacientes con EC no se ha estudiado previamente.
Nuestro objetivo fue analizar la microbiota fecal de pacientes estreñidos antes y después de consumir un probiótico de una sola cepa (B. infantis cepa 35624).
Material y métodosUsamos secuenciación de alto rendimiento del gen 16S ARNr para analizar la microbiota fecal de pacientes mujeres (n=13) con EC. Se indicó a los pacientes que consumieran una cápsula de Alflorex® (con 1×109UFC/g de B. infantis 35624) al día durante 8 semanas. Se obtuvieron muestras fecales al inicio del estudio y al final de la administración de probióticos.
ResultadosLas métricas de diversidad alfa no fueron diferentes entre la línea de base y la final. El productor de butirato Oscillospira fue el taxón más fuertemente correlacionado con las variantes de secuencia de amplicones (R2=0.55, p<0.0001). Excepto por unos pocos taxones bacterianos, no hubo diferencias significativas en la abundancia relativa entre la línea de base y la final. Las medidas de diversidad beta también mostraron evidencia limitada de diferencias entre los dos periodos de tiempo.
ConclusionesLos resultados sugieren que la microbiota bacteriana fecal permanece estable en mujeres estreñidas que consumen un probiótico de una sola cepa. Estos hallazgos pueden ser útiles para comprender mejor el funcionamiento de los probióticos en pacientes con trastornos digestivos.
In recent years, probiotics have been used in functional gastrointestinal disorders, including chronic constipation (CC). The effect of Bifidobacterium infantis strain 35624 on the gut microbiota of CC patients has not been previously studied.
Our aim was to analyze the fecal microbiota of constipated patients, before and after consuming a single-strain probiotic (B. infantis strain 35624).
Material and methodsWe used 16S rRNA gene high-throughput sequencing to analyze the fecal microbiota of female patients (n=13) with CC. Patients were instructed to ingest one capsule of Alflorex® (containing 1×109CFUs/g B. infantis strain 35624) daily for eight weeks. Fecal samples were obtained at the baseline and end (final) of probiotic administration.
ResultsAlpha diversity metrics did not differ between the baseline and final periods. The butyrate producer, Oscillospira, was the taxon most strongly correlated with amplicon sequence variants (R2=0.55, p<0.0001). Except for a few bacterial taxa, there were no significant differences in relative abundance between the baseline and final periods. Beta-diversity measures also showed limited evidence for the differences between the two time periods.
ConclusionsThe results suggest that the fecal bacterial microbiota remains stable in constipated women consuming a single-strain probiotic. Those findings may be helpful in better understanding probiotic functioning in patients with digestive disorders.
El tubo digestivo humano es hogar de millones de microorganismos altamente competitivos (conocidos como la microbiota intestinal) que viven en íntima simbiosis con el anfitrión1. La microbiota intestinal juega un papel vital en la regulación y mantenimiento de la salud en el tubo digestivo y en órganos extraintestinales2. Es importante investigar las características de dicha simbiosis para comprender mejor la salud y el bienestar humanos.
Los primeros estudios que investigaron a los microbios en intestinos y en otros órganos mostraron que existía una gran variación entre individuos sanos, pero que permanecían relativamente estables durante periodos de tiempo cortos3. Sin embargo, otros estudios sugieren una variabilidad temporal pronunciada en el microbioma humano sano, incluso de un día a otro4. Esta variación es particularmente relevante en contextos clínicos, por ejemplo, en pacientes con trastornos gastrointestinales o en quienes reciben antibióticos5,6.
El estreñimiento crónico (EC) es un trastorno intestinal de alta prevalencia que se caracteriza por heces duras y/o una sensación de evacuación incompleta7. Se recomiendan diferentes tratamientos para aliviar dichos síntomas, incluido el uso de probióticos (es decir, microorganismos vivos benéficos). Algunos mecanismos por medio de los que los probióticos pueden mejorar la salud en pacientes estreñidos incluyen la alteración de la microbiota intestinal y la regulación del medio ambiente intestinal, como la reducción del pH luminal. Varios ensayos aleatorizados han sido realizados con el uso de probióticos (principalmente con cepas de Lactobacillus y Bifidobacterium) en EC7, con éxito clínico variado. Un metaanálisis reciente de ensayos aleatorizados concluyó que el consumo de probióticos puede reducir el tiempo de tránsito intestinal, incrementar la frecuencia de heces y mejorar la consistencia de las heces en adultos con estreñimiento funcional8. Sin embargo, los autores también han reportado una amplia variación de desempeño entre los ensayos. La razón para dicha variación podría involucrar diferencias entre las poblaciones de pacientes, al igual que en el desempeño de los probióticos9.
El uso de la Bifidobacterium como probiótico está bien documentado en pacientes con trastornos gastrointestinales funcionales, pero también ha mostrado resultados diversos. Dimidi et al.10 reportaron que una suplementación por 4 semanas de 1.5×1010 de unidades formadoras de colonias (UFC) de una cepa de B. lactis no fue efectiva para el manejo del EC, en un ensayo aleatorizado con 75 participantes. En ese estudio, los autores analizaron la microbiota intestinal, pero solo utilizaron PCR en tiempo real cuantitativo (PCRc), lo cual es útil para monitorear taxones individuales, pero no es capaz de caracterizar completamente a comunidades microbianas. Otro estudio investigó el efecto de la suplementación de 8 semanas de 1×109UFC de cepa 35624 de B. infantis en la microbiota fecal de pacientes con síndrome de intestino irritable (SII)11. El estudio mostró mayor excreción del probiótico administrado durante el tratamiento, desapareciendo poco después de la suspensión de la administración. Sin embargo, los autores también utilizaron PCRc solo para algunos taxones seleccionados, y concluyeron de manera correcta que dada la complejidad del espectro de la microbiota intestinal, los cambios o diferencias en los taxones seleccionados no fueron indicadores útiles de la función probiótica. El objetivo del presente estudio fue analizar la microbiota fecal de pacientes estreñidos, previo y posterior al consumo del probiótico de una sola cepa, B. infantis 35624.
Materiales y métodosReclutamiento de pacientesReclutamos a pacientes con EC del puerto de Veracruz, México, para el presente ensayo abierto. Los criterios de inclusión fueron los siguientes: >18 años de edad, autorreporte de frecuencia de heces de 3 movimientos intestinales por semana o menos, autorreporte de consistencia de heces de tipo 1-4 en la Escala de Heces de Bristol, y cumplir con los criterios de diagnóstico de Roma IV para estreñimiento funcional12,13.
A los pacientes que estaban recibiendo tratamiento farmacológico se les solicitó que interrumpieran el tratamiento por al menos una semana, para poder ser evaluados. Los criterios de exclusión fueron los siguientes: trastornos gastrointestinales y otros que pudieran afectar la motilidad intestinal, cirugía abdominal previa, terapia activa con medicamentos conocidos por afectar la motilidad intestinal, causas secundarias de estreñimiento, defecación disinérgica, inercia colónica, problemas anorrectales, intolerancia a la lactosa, alergia a leche de vaca y soya, consumo regular de probióticos, prebióticos, suplementos de fibra y laxantes, uso de antibióticos hasta 4 semanas antes del inicio del ensayo, abuso activo de alcohol, drogas o medicamentos. La prevalencia del estreñimiento es casi dos veces más común en mujeres, por lo que decidimos incluir solo a mujeres, para evitar factores potencialmente confusos.
Se les indicó a los pacientes ingerir una cápsula diaria de Alflorex®, con 1×109 de UFC/g de B. infantis cepa 35624, por un periodo consecutivo de 8 semanas, manteniendo sus hábitos alimenticios regulares. La B. infantis 35624 también es conocida como B. longum subespecie infantis (https://www.precisionbiotics.com/es/), pero además, ha sido catalogada (de manera más precisa) como B. longum subespecie longum (el error en asignación original fue resultado del uso único de la clasificación taxonómica basada en 16S ARNr parcial)14. El probiótico fue donado por la compañía Menarini México (Ciudad de México, México), la cual solo proporcionó el fármaco y no intervino en el diseño del estudio o en el análisis de los resultados.
Se les informó a los pacientes que estaban siendo invitados a participar en un estudio cuyo objetivo era investigar el efecto de un probiótico sobre sus síntomas y la composición de la microbiota intestinal, sin prometer de manera explícita un efecto benéfico. Considerando la cantidad de producto en cada cápsula (247mg) y la concentración del probiótico (1×109UFC/g), cada cápsula proporcionó 2.47×108UFC. Los pacientes respondieron un cuestionario respecto a parámetros clínicos como consistencia de heces, percepción de intensidad de sensación de distensión y días sin síntomas, en línea de base y al final de la administración de los probióticos. La consistencia de heces fue evaluada por los pacientes utilizando la Escala de Heces de Bristol15, en la cual el 1 representa trozos duros y el 7 representa heces aguadas. La percepción de la intensidad de sensación de distensión fue expresada utilizando una escala visual análoga de 0 a 100. Se evaluó la mejoría global, utilizando la pregunta general de síntomas: «En los últimos 7 días, ¿consideras que tuviste un alivio adecuado de tus síntomas de estreñimiento?» Se les dio instrucciones a los pacientes de usar 17g diarios de polietilenglicol, de forma regular de ser necesario, si se requería terapia de rescate.
Recolección de muestras fecales y extracción de ADNLas muestras fecales fueron recolectadas por los pacientes en sus casas, en línea de base y al final de la administración de probióticos. Las muestras fueron colocadas en hieleras con paquetes de gel congelados y llevadas a nuestro laboratorio en el lapso de una hora, donde fueron colocadas a −20°C, hasta ser procesadas. Debido a que la homogenización de las heces evita una evaluación precisa de las comunidades microbianas16, descongelamos las muestras y obtuvimos las alícuotas para el análisis, principalmente de la parte media del material fecal disponible de cada muestra17. El ADN genómico total fue obtenido y purificado de una alícuota (100mg) de cada muestra fecal, utilizando centrifugación para la disrupción celular, en conjunto con un kit comercial de extracción de ADN (Wizard® Genomic DNA Purification Kit Protocol, Promega Corporation, Madison, Wisconsin, EE.UU.). Se procesaron dos alícuotas separadas de la muestra de línea de base del último paciente (ID de muestra: S19) para evaluar la replicabilidad del procedimiento. Las muestras de ADN (n=27, 26 de 13 pacientes más un duplicado de S19) fueron ajustadas a 150ng/μl y enviadas a Molecular Research LP (Shallowater, Tejas, EE.UU.). Se realizó una PCR para amplificar una región semiconservada del gen 16s ARNr utilizando los cebadores 515F (GTGYCAGCMGCCGCGGTAA) y 806R (GGACTACNVGGGTWTCTAAT). La secuenciación de alto rendimiento de ADN de los amplicones de PCR fue realizada en un instrumento MiSeq (Illumina, San Diego, California, EE.UU.), de la manera descrita en otra instancia18. A menos que se declare de otra manera, solo los resultados de los 13 pares de muestras (n=26), usando la primera réplica S19, se muestran y discuten.
Bioinformática y estadísticasUtilizamos diferentes herramientas en QIIME219 v.2020.2 para analizar los datos de secuenciación, usando los parámetros por defecto, a menos que se especifique de otra manera. El laboratorio de secuenciación proporcionó asignación taxonómica libre, utilizando una base de datos curada, pero los resultados solo se mencionan en el contexto de nuestra asignación taxonómica en QIIME2, usando la base de datos Greengenes 13.8. El método filogenético UniFrac20 fue utilizado para comparar comunidades microbianas, con distancias ponderadas y sin ponderar21.
Es práctica común analizar los diferentes taxones bacterianos por separado. Sin embargo, este abordaje no es necesariamente el mejor para comprender las fluctuaciones microbianas en ecosistemas complejos, donde coexisten diferentes tipos y cantidades de microbios. Por lo tanto, usamos el SAS® University Edition (SAS Institute, Cary, Carolina del Norte, EE.UU.) para realizar el análisis multivariado para diferentes taxones al mismo tiempo. En los casos necesarios se usó la prueba de Wilcoxon de rango con signo para comparar variables (p.ej., abundancia relativa de bacterias) entre ambos periodos de tiempo (de línea de base y final). También realizamos análisis de correlación utilizando los valores de línea base y final para los taxones individuales, al igual que entre las diferentes combinaciones de los puntos de tiempo y taxones (p.ej., valores de línea base para taxón A vs. valores finales para taxón B), para investigar cualquier relación que pudiera impactar la variabilidad de microbios a lo largo del tiempo. Las gráficas fueron generadas utilizando SAS y PAST v.3.25.
Consideraciones éticasSe requirió de un consentimiento informado por escrito para la participación en el presente estudio. Todos los procedimientos fueron realizados de acuerdo con el código de ética de la OMS (Declaración de Helsinki) y aprobados por el comité de ética local del Instituto para la Investigación Biológica Médica (Universidad Veracruzana, 2018-013R). Los autores declaran que el presente artículo no contiene información que permita identificar a los pacientes.
ResultadosPacientesSe incluyó a 19 pacientes mujeres, pero solamente 13 pudieron completar el protocolo (tabla 1). Previo al estudio, 3 pacientes no estaban tomando medicamento o suplemento para tratar su estreñimiento, 6 tomaban suplementos de fibra y 11 usaban laxantes (6 polietilenglicol, 3 lactulosa).
Características de línea de base de las 13 pacientes que completaron el protocolo
| ID de sujeto | Rango de edad | Índice de masa corporal | Porcentaje autopercibido de intensidad de distensión (línea de base/final) |
|---|---|---|---|
| S01a | 50-60 | 27.3 | 50/0 |
| S02a | 50-60 | 25.5 | 100/25 |
| S06a | Más de 60 | 28.2 | 75/0 |
| S07a | Más de 60 | 25.2 | 75/50 |
| S08a | 30-40 | 33.7 | 100/90 |
| S09a | 30-40 | 25.9 | 75/50 |
| S10 | 50-60 | 25.5 | 25/50 |
| S11 | 40-50 | 25.5 | 65/75 |
| S14 | 40-50 | 27.1 | 50/NR |
| S15 | 50-60 | 20.0 | 25/25 |
| S16a | 50-60 | 25.2 | 75/25 |
| S18a | 50-60 | 37.6 | 50/10 |
| S19 | 40-50 | 29.7 | 100/100 |
NR: no registrado y en consecuencia no incluido en la comparación estadística.
De las 13 pacientes reclutadas, 6 (46%) reportaron una mejoría en la Escala de Heces de Bristol de más del 50%, en comparación con la línea de base, y 10 (77%) de las 13 participantes reportaron una mejoría de síntomas en general. La intensidad de la sensación de distensión fue significativamente más baja después de la administración de probióticos (67% vs. 42%, p=0.02) (tabla 2) y el porcentaje de tiempo (en días) sin síntomas también fue más bajo posteriormente al probiótico (49% vs. 39%), pero la diferencia no fue significativa (p=0.48). Solo 2 pacientes reportaron haber tomado el medicamento de rescate, de manera intermitente. Sin embargo, es probable que estos resultados tengan relevancia clínica limitada debido a un posible efecto placebo y a la falta de un grupo control.
Proporciones relativas promedio de las secuencias del gen 16S ARNr para algunos de los taxones más abundantes
| Taxón | Línea de base | Final | Valor de p | Pacientes que experimentaron aumento/disminución |
|---|---|---|---|---|
| Firmicutes | 66.8% | 54.2% | 0.28 | 6 (16.4%)/7 (35.6%)* |
| Blautia | 14.0% | 7.2% | 0.10 | 5 (5.8%)/8 (14.7%) |
| Clostridium | 8.4% | 8.9% | 0.86 | 6 (10.8%)/7 (8.1%) |
| Eubacterium | 6.9% | 6.3% | 0.51 | 4 (4.4%)/9 (2.8%) |
| Ruminococcus | 6.6% | 5.7% | 0.46 | 4 (7.6%)/9 (4.7%) |
| Turicibacter | 9.1% | 3.3% | 0.46 | 7 (4.2%)/6 (17.4%) |
| Bacteroidetes | 12.2% | 18.3% | 0.38 | 8 (17.6%)/5 (12.3%) |
| Bacteroides | 3.5% | 6.1% | 0.65 | 9 (6.9%)/4 (6.9%) |
| Prevotella | 3.9% | 5.2% | 0.75 | 7 (5.9%)/6 (4.3%) |
| Cianobacteria | 10.9% | 9.9% | 0.92 | 7 (8.6%)/6 (11.9%) |
| Halospirulina | 10.8% | 9.9% | 0.92 | 7 (8.6%)/6 (11.9%) |
| Proteobacteria | 3.2% | 10.1% | 0.01 | 11 (8.5%)/2 (2.5%) |
| Alfaproteobacteria | 0.5% | 2.1% | 0.70 | 7 (3.7%)/6 (0.8%) |
| Betaproteobacteria | 0.9% | 0.4% | 0.70 | 8 (0.6%)/5 (2.3%) |
| Deltaproteobacteria | 0.01% | 1.3% | 0.02 | 10 (1.6%)/3 (0.005%) |
| Epsilonbacteria | 0.007% | 0.6% | 0.03 | 9 (0.8%)/4 (0.003%) |
| Gammaproteobacteria | 1.8% | 5.7% | 0.03 | 10 (5.5%)/3 (1.6%) |
| Stenotrophomonas | 1.4% | 4.2% | 0.60 | 7 (6.3%)/6 (1.1%) |
| Kopriimonas | 0.2% | 1.9% | 0.92 | 6 (3.9%)/7 (0.3%) |
| Pseudomonas | 0.02% | 0.7% | 0.02 | 10 (0.8%)/3 (0.02%) |
| Actinobacteria | 6.1% | 3.9% | 0.42 | 6 (2.1%)/7 (5.8%) |
| Bifidobacterium | 4.2% | 2.7% | 0.42 | 5 (1.3%)/8 (3.3%) |
| Verrucomicrobia | 0.8% | 3.4% | 0.17 | 8 (4.7%)/5 (0.5%) |
| Akkermansia | 0.7% | 3.4% | 0.10 | 8 (4.7%)/5 (0.5%) |
Los taxones están organizados por filo (destacados en negrita para auxiliar la visualización). Los valores de p provienen de la prueba de Wilcoxon.
Después de la demultiplexación, obtuvimos 2,069,625 secuencias 16S de 27 muestras fecales (media: 76,652 secuencias/muestra, mín.: 25,964, máx.: 170,946). La utilización de DADA222 para el filtrado de calidad resultó en 1,868,215 secuencias (∼90% del original), utilizando 280 nucleótidos como longitud de truncado.
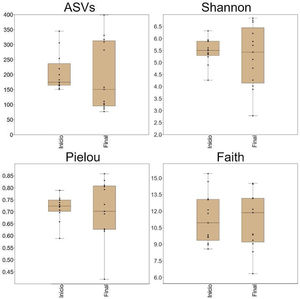
Diversidad alfaUsando una profundidad de rarefacción de 24,000 secuencias/muestra, DADA2 reveló la presencia de 1,123 variantes de secuencia de amplicones (VSA) que pertenecieron a 120 géneros de 16 filos. No existieron diferencias significativas en el número de VSA, valores de diversidad Shannon, valores de uniformidad de Pielou y los valores de diversidad filogenética de Faith (fig. 1). Con la excepción de los valores de diversidad filogenética de Faith, existió una disimilitud notable en la dispersión de las muestras, entre la línea de base y los periodos finales (fig. 1).
Diagramas de aja con jitter que muestran métricas de diversidad alfa. No hubo diferencia estadísticamente significativa entre inicio y final, pero los valores de la mayoría de las métricas mostraron una mayor variación después del probiótico. Los diagramas se construyeron en PAST v.3.25. ASVs: variantes de secuencia de amplicón.
Un total de 99.3% de las lecturas del gen 16S podrían ser clasificadas en un filo dado, pero solo el 63% fueron asignadas a un género. Este porcentaje es común23, pero la mayoría de los artículos de investigación prefiere no declarar los porcentajes de lecturas asignados a los diferentes niveles taxonómicos. Siete filos sumaron el ∼99% de todas las lecturas (Firmicutes: 55.8%, Bacteroidetes: 15.0%, Cianobacteria: 10.2%, Tenericutes: 5.4%, Proteobacteria: 5.3%, Actinobacteria: 4.9% y Verrucomicrobia: 2.0%). Estos resultados concuerdan parcialmente con los resultados proporcionados por el laboratorio de secuenciación que utiliza una base de datos diferente para las asignaciones taxonómicas (Firmicutes: 60.6%, Bacteroidetes: 15.5%, Cianobacteria: 10.3%, Proteobacteria: 6.5%, Actinobacteria: 4.9% y Verrucomicrobia: 2.0%), con la excepción de Tenericutes, que solo sumó el 0.003% de todas las lecturas. Con pocas excepciones, la variación en abundancia relativa de taxones entre dos réplicas de la línea de base de la muestra S19 fue menor en comparación con la variación entre la línea de base y los valores finales de todos los pares de muestras.
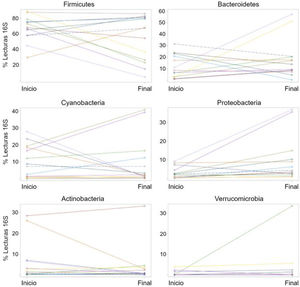
La abundancia relativa de cada filo reveló una variación interindividual alta, al igual que una amplia diferencia en la variación a lo largo del tiempo para cada sujeto (fig. 2), como se muestra en otra instancia. Por ejemplo, 7 de las 13 pacientes (54%) experimentaron una disminución en la abundancia relativa de Firmicutes de 37% en promedio, mientras que 6 de las 13 pacientes (46%) mostraron un incremento en el taxón de 16%, en promedio (tabla 2). En otras palabras, el cambio en Firmicutes a lo largo del tiempo sucedió en una proporción similar de pacientes (54% vs. 46%), pero el cambio fue más pronunciado (disminución de 37% vs. aumento de 16%) en los pacientes que experimentaron una disminución en dicho taxón. Independientemente de esto, la diferencia en Firmicutes entre los dos periodos de tiempo no llegó a la significación estadística (p=0.28). En el caso de Bacteroidetes, 5 de las 14 pacientes (38%) mostraron una disminución de 12% en el taxón, en promedio, mientras que 8 de las 13 pacientes (62%) mostraron un incremento de 18%, en promedio (tabla 2). La diferencia en Bacteroidetes entre los dos puntos temporales no llegó tampoco a la significación estadística (p=0.38) (tabla 2). Dos pacientes mostraron una pequeña disminución en Proteobacteria (de 7.4% a 2.6%, y de 1.97% a 1.94%, respectivamente) posteriormente a la administración del probiótico, mientras que otras 11 pacientes mostraron un aumento promedio de 8.5% en el taxón (fig. 2). La diferencia en Proteobacteria entre los periodos de línea de base (3.2%) y final (10.1%) fue estadísticamente significativa (p=0.01), aunque fue principalmente causada por dos participantes (fig. 2) y no estuvo claramente asociada con ningún taxón en particular (tabla 2).
Porcentajes de lecturas del gen 16S rRNA a nivel de filo para los 13 pacientes al inicio y al final de la administración del probiótico. El propósito de esta figura es ayudar a visualizar la alta variación interindividual (separación de puntos dentro de cada punto de tiempo), así como la gran diferencia en la variación a lo largo del tiempo (cambio en las lecturas de 16S del inicio al final) para cada sujeto.
Las gráficas fueron construidas en SAS.
La abundancia relativa del género Bifidobacterium fue relativamente alta (el décimo género más abundante) en nuestro análisis en QIIME2 (promedio 3.4%, mín.: 0.07%, máx.: 31.5%) y en los resultados del laboratorio de secuenciación (promedio 3.3%, mín.: 0.05%, máx.: 30.8%).
Observamos 9 VSA que estuvieron relacionadas con Bifidobacterium, pero la mayoría fueron bajas en abundancia y mostraron baja prevalencia (tabla 3).
Abundancia relativa y prevalencia de las 9 VSA de Bifidobacterium
| Taxonomía de VSA con similitud de porcentajes | Línea de base | Final | Valor de p |
|---|---|---|---|
| B. adolescentis (98.8%) | 2.1% (13/13) | 1.4% (13/13) | 0.38 |
| B. adolescentis (99.3%) | 1.5% (13/13) | 1.1% (13/13) | 0.46 |
| B. longum (100%) | 0.6% (13/13) | 0.2% (12/13) | 0.08 |
| B. adolescentis (99.2%) | 0.07% (12/13) | 0.05% (11/13) | 0.81 |
| B. adolescentis (99.2%) | 0.01% (7/13) | 0.008% (6/13) | NA |
| B. breve (69.6%) | 0.0004% (2/13) | 0.002% (1/13) | NA |
| B. adolescentis (95.7%) | 0.0002% (2/13) | 0.004% (1/13) | NA |
| B. adolescentis (94.5%) | 0.0002% (2/13) | 0.0002% (2/13) | NA |
| B. breve (69.6%) | 0.003% (1/13) | 0.003% (1/13) | NA |
Los valores de p provienen de la prueba de Wilcoxon.
NA: la comparación no aplica; VSA: variantes de secuencia de amplicones.
Utilizamos la abundancia relativa de las lecturas del 16S de 6 de los filos más abundantes, en diferentes combinaciones, para encontrar el valor más bajo de la lambda de Wilks. En consecuencia, la combinación de 3 filos (Firmicutes, Proteobacteria y Verrucomicrobia) fue asociada con el valor más bajo (tabla suplementaria S1). Esto es interesante porque implica que la combinación de esos 3 taxones puede ser más precisa para analizar las diferencias entre los dos puntos temporales.
Análisis de correlaciónLa abundancia relativa de Actinobacteria (R2=0.5, p=0.007) presentó la correlación más fuerte entre los periodos de línea de base y final, pero esto fue causado por los datos de un paciente (tabla suplementaria S2). El único otro filo que mostró una tendencia a la significación fue la Proteobacteria (R2=0.3, p=0.046, tabla suplementaria S2). También examinamos las correlaciones entre los diferentes filos en los diferentes puntos temporales (tabla suplementaria S3). De manera interesante, existió una correlación inversa entre los valores de línea de base de Firmicutes y Proteobacteria (R2=0.73, p=0.0002), como se mostró recientemente en un estudio sobre la microbiota en leche24. Otro hallazgo interesante fue la correlación positiva entre la línea de base de Cianobacteria y Proteobacteria (R2=0.82, p=2.4×10-5). Las cianobacterias son frecuentemente consideradas contaminantes, pero Ley et al.25 propusieron la interesante posibilidad de que las cianobacterias intestinales sean descendientes de cianobacterias no fotosintéticas ancestrales que se adaptaron a la vida dentro del tubo digestivo.
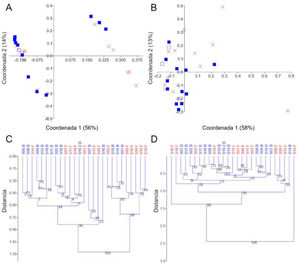
Diversidad betaLa utilización de PCoA y agrupamiento UPGMA en distancias UniFrac no ponderadas sugirió poca variación en las dos réplicas del S19 y la presencia de dos grupos de muestras claros que se explicaron parcialmente por la administración del probiótico (fig. 3A y C). Debido a la posible existencia de enterotipos (grupos de muestras identificables por la variación en la presencia de taxones específicos y sus niveles26), retiramos los diferentes taxones, uno por uno, para investigar si la ausencia de un taxón dado explicaría dicho agrupamiento. Sin embargo, no encontramos evidencia que sugiera que el agrupamiento fue debido a la presencia (o ausencia) de un taxón en particular. De manera interesante, los dos grupos de muestras desaparecieron al analizar los datos de grupos separados de taxones seleccionados (p.ej., Firmicutes, Proteobacteria y Verrucomicrobia), pero la significación de este hallazgo aún no es clara para nosotros. A diferencia de los resultados sobre las distancias UniFrac no ponderadas, el análisis de las distancias UniFrac ponderadas mostró una separación de réplicas de S19 más fuerte, pero no reveló un agrupamiento significativo de muestras (fig. 3B y D). Independientemente de cualquier agrupamiento visual, la prueba PERMANOVA no mostró diferencias significativas en las distancias UniFrac no ponderadas (p=0.12) o ponderadas (p=0.81).
Gráficos PCoA y árboles UPGMA utilizando distancias UniFrac no ponderadas y ponderadas. Gráficos PCoA usando distancias no ponderadas (A) y ponderadas (B), árboles UPGMA usando distancias no ponderadas (C) y ponderadas (D). En A y B, las muestras de línea de base están representadas por cuadrados azules llenos y las muestras finales por una X roja. La muestra que se procesó por duplicado (S19, línea de base) se muestra con un símbolo diferente (cuadrados azules vacíos) para una mejor visualización. Los árboles se construyeron utilizando 10,000 muestras de arranque. El índice de similitud Euclidiana se utilizó para todas las gráficas en PAST v. 3.25.
El índice de masa corporal y la edad fueron las únicas variables continuas asociadas con los pacientes y no explicaron significativamente el número de VSA observado. Algunos de los taxones estuvieron relacionados con el número de VSA, en particular el productor de butirato, Oscillospira (R2=0.55, p<0.0001), un miembro enigmático de Firmicutes (familia Ruminococcaceae) de la microbiota intestinal humana que ha sido demostrado que tiene una asociación positiva con delgadez y salud27 (tabla suplementaria S4).
Microbioma centralEl concepto de microbioma central se refiere a las especies (p.ej., VSA) que están presentes en la mayoría de las muestras en un medio ambiente dado, por ejemplo, las heces humanas. Pocas unidades taxonómicas operacionales (UTO) fueron compartidas por el 50% de las muestras (122 de 1,123, o 10.9% de todas las VSA), de línea de base (153, 19.1%) o del final (105, 12.3%), lo que implica que la mayoría de las VSA fueron singulares para cada individuo.
DiscusiónLa microbiota intestinal es relevante en la patogénesis y manejo de trastornos gastrointestinales, incluido el estreñimiento28. En México, el EC tiene una prevalencia de entre 2.4% y 22.3%29, lo cual representa aproximadamente 3-27 millones de personas. Aunque algunos estudios han reportado resultados favorables para los probióticos en el estreñimiento, su eficacia clínica para aliviar los síntomas es aún controvertida, ya que existen diferencias en los abordajes metodológicos, como las poblaciones de estudio (niños, adultos, adultos mayores) y productos probióticos (probióticos de una sola especie o de múltiples especies). Además, solo pocos estudios han explorado el impacto de los probióticos sobre la composición y estabilidad de la microbiota intestinal en pacientes estreñidos. El presente estudio investigó la microbiota fecal de pacientes mujeres con EC, después de la administración de un probiótico comercial de una sola cepa.
Las heces de pacientes estreñidos a menudo parecen diferir respecto al contenido de agua, en comparación con las heces de individuos sanos, y el cambio físico en la consistencia de las heces puede en sí mismo tener un impacto en la composición y funcionamiento de la microbiota intestinal30. Sin embargo, la consistencia de las heces es difícil de contextualizar con la efectividad del probiótico debido a variaciones en el tamaño de las células y el volumen de la microbiota intestinal, lo cual es un tema que no se ha discutido adecuadamente en la literatura. Levin y Angert31 reportaron que el volumen común de bacterias tiene un rango de 0.4 a 3μm3(o 0.4 a 3 femtolitros o fl; 1ml=1×1012fl). Considerando que 1ml de heces húmedas pesa aproximadamente 1g, entonces cada mg de heces húmedas equivale a 1×109fl. Si consideramos que las células bacterianas constituyen aproximadamente 150mg de cada gramo de heces32, y consideramos los rangos de volumen mencionados antes, implica que cada gramo de heces húmedas en individuos sanos puede contener de 5×1010 (considerando un volumen de 3fl por cada célula bacteriana) a 3.8×1011 (considerando un volumen de 0.4fl) células bacterianas. En caso del 100% de supervivencia de todos los probióticos en estómago e intestino delgado, la microbiota intestinal colónica de cada paciente del presente estudio recibiría una dosis diaria de 2.47×108UFC, o aproximadamente 100 veces menos células bacterianas que en 1g de heces. En caso de cambio de consistencia en heces (p.ej., utilizando la Escala de Heces de Bristol), es entonces plausible asumir que el número total de células bacterianas autóctonas por gramo de contenido cambiaría, lo cual podría afectar la bioquímica de la microbiota intestinal. En general, el estreñimiento es una condición clínica que puede afectar la densidad microbiana, y el cambio resultante podría tener otras implicaciones importantes sobre la salud. Este es un tema importante que no se ha explorado y amerita más investigaciones.
Además de las preocupaciones sobre la consistencia de heces discutidas anteriormente, existen otros aspectos desconocidos respecto al efecto de los probióticos in vivo, por ejemplo, la interacción con el microbioma comensal residente9 y otras condiciones específicas que se encuentran cada día de consumo de probióticos. Por ejemplo, Vitali et al.33 sugirieron que la colonización intestinal por bacterias probióticas podría ser más alta en sujetos que tienen menor concentración de esas bacterias, previamente a la ingesta de probióticos, mientras que otros han sugerido lo opuesto34. En el presente estudio, no encontramos relaciones significativas en la abundancia relativa microbiana entre los periodos de línea de base y final, aunque esto podría ser debido al largo periodo (clínicamente indicado) de tiempo de administración del probiótico. Sin embargo, la diferente distribución de los valores de uniformidad de Pielou y otras métricas de diversidad alfa, posterior a la administración de probiótico, es interesante porque otros estudios han mostrado una distribución similar de dichos valores, incluso en medios ambientes drásticamente diferentes, como los intestinos y la lengua (tutorial de QIIME2 «imágenes en movimiento» de los datos publicados por Caporaso et al.)4.
El término especies es evidentemente impreciso para clasificar a las bacterias en grupos, y se han realizado varios esfuerzos por llegar a un consenso sobre nombres y clasificaciones de bacterias35. Esta búsqueda de una identificación precisa de las especies, subespecies y cepas bacterianas es particularmente relevante con relación al etiquetado de productos comerciales que contienen probióticos, por ejemplo, miembros de las Bifidobacterium36,37. Esto es importante ya que los efectos benéficos de los probióticos son específicos a las cepas, por lo tanto, no todos los miembros de una misma especie comparten las mismas propiedades. Bifidobacterium es un taxón bacteriano que llamó la atención en las últimas décadas del siglo XX38. Varios estudios han utilizado diferentes especies y cepas de Bifidobacterium como probióticos39,40, pero los contenidos descritos en las etiquetas de productos comerciales frecuentemente son imprecisos respecto a los tipos y cantidades de microorganismos41. Para superar algunas de estas inconsistencias, decidimos usar un producto comercial que ha sido sometido a un riguroso control de calidad y que contiene una cepa bien estudiada de Bifidobacterium, que ha demostrado ejercer efectos inmunorreguladores42,43.
El mecanismo de acción de los probióticos en pacientes con enfermedades gastrointestinales es un fenómeno complejo de investigar. Varios ensayos aleatorizados, controlados con placebo, han demostrado mejoría en síntomas de SII, incluido el estreñimiento, después de la ingesta de B. infantis 35624 y otros probióticos, pero pocos estudios han explorado la estabilidad a lo largo del tiempo, o discutido los posibles modos de acción44,45. Falony et al.46 mostraron una alimentación cruzada in vitro entre una cepa de B. longum y bacterias del colon productoras de butirato y conversoras de acetato, durante el crecimiento en oligofructosa. Fukuda et al.47 expandieron esos resultados y demostraron que algunas cepas de Bifidobacterium pueden proteger contra infecciones enteropatogénicas por medio de la producción de acetato, lo cual puede contribuir a la formación de butirato48. Este cambio en la bioquímica intestinal se relaciona finalmente con una modulación de la composición de la microbiota intestinal, lo cual puede ser evidenciado por cambios en las proporciones relativas de microorganismos fecales, posteriores a la suplementación de probióticos. Sin embargo, nuestros resultados sugieren que el consumo de B. infantis 35624 no está relacionado con cambios en las proporciones generales de bacterias, al menos en muestras fecales. El caso de la Proteobacteria es de particular interés porque ese taxón mostró una abundancia relativa significativamente diferente entre los periodos de línea de base y final. Aunque la Proteobacteria se asocia frecuentemente con efectos negativos sobre la salud, nosotros y otros autores hemos demostrado que algunos taxones dentro de la Proteobacteria podrían ser benéficos, como la Pseudomonas18. Estos resultados agregan información valiosa a la literatura sobre probióticos, en particular en el contexto de interacciones microbianas en los intestinos de pacientes estreñidos, y enfatiza que el beneficio potencial de los probióticos es más complejo que un simple cambio en las cantidades y proporciones de bacterias.
Las limitaciones de nuestro estudio incluyen la falta de un grupo placebo y la posibilidad de que algunos de los pacientes no hayan sido regulares con la ingesta del producto. Se registró la respuesta clínica autopercibida de la administración del probiótico, pero el hecho de que el efecto placebo sea notoriamente amplio, particularmente en ensayos abiertos que involucran a pacientes con trastornos intestinales funcionales, debe ser considerado. Adicionalmente, cada persona no solo lleva una dieta diferente, sino que también un patrón alimentario que obedece a preferencias personales (p.ej., comida casera vs. comida comercial) y otros factores (p. ej., compromisos laborales), y la adherencia al tratamiento no fue evaluada rigurosamente. Finalmente, en el presente ensayo utilizamos muestras fecales que no necesariamente reflejan cambios biológicamente significativos en la microbiota asociada a la mucosa, y solo utilizamos secuenciación de alto rendimiento para estudiar la microbiota. Otras técnicas moleculares, como la PCRc, podrían haber confirmado la falta de cambios significativos para taxones específicos.
ConclusionesEl presente estudio mostró pequeñas diferencias en la microbiota fecal y varios parámetros de diversidad, previa y posteriormente a la administración de un probiótico de una sola cepa en pacientes estreñidas femeninas. Aunque se pueden extraer conclusiones limitadas de este estudio, los resultados contribuyen a la mejor comprensión del funcionamiento de los probióticos en pacientes estreñidos que consumen fórmulas de probióticos de una sola cepa.
FinanciaciónNo se recibió patrocinio de ningún tipo para llevar a cabo este artículo.
Conflicto de interesesJ.M.R.-T. ha recibido honorarios como conferencista, viáticos, y participado en reuniones médicas de mesa directiva con Takeda México, Asofarma y Menarini. EMMQ ha recibido honorarios por consultoría de Novo-zymes, honorarios como conferencista de Biocodex y apoyo para investigación de 4D pharma. El resto de los autores declara no tener conflicto de intereses.