La microbiota intestinal es la comunidad de microorganismos vivos residentes en el tubo digestivo. Muchos grupos de investigadores a nivel mundial trabajan descifrando el genoma de la microbiota. Las técnicas modernas de estudio de la microbiota nos han acercado al conocimiento de un número importante de bacterias que no son cultivables, y de la relación entre los microorganismos que nos habitan y nuestra homeostasis. La microbiota es indispensable para el correcto crecimiento corporal, el desarrollo de la inmunidad y la nutrición. Las alteraciones en la microbiota podrían explicar, por lo menos en parte, algunas epidemias de la humanidad como el asma y la obesidad. La disbiosis se ha asociado a una serie de trastornos gastrointestinales que incluyen el hígado graso no alcohólico, la enfermedad celíaca y el síndrome de intestino irritable. En el presente trabajo trataremos sobre la nomenclatura, las técnicas de estudio modernas, las funciones de la microbiota intestinal y la relación que tiene con la salud y la enfermedad.
Gut microbiota is the community of live microorganisms residing in the digestive tract. There are many groups of researchers worldwide that are working at deciphering the collective genome of the human microbiota. Modern techniques for studying the microbiota have made us aware of an important number of nonculturable bacteria and of the relation between the microorganisms that live inside us and our homeostasis. The microbiota is essential for correct body growth, the development of immunity, and nutrition. Certain epidemics affecting humanity such as asthma and obesity may possibly be explained, at least partially, by alterations in the microbiota. Dysbiosis has been associated with a series of gastrointestinal disorders that include non-alcoholic fatty liver disease, celiac disease, and irritable bowel syndrome. The present article deals with the nomenclature, modern study techniques, and functions of gut microbiota, and its relation to health and disease.
Lo que sabemos de la interesante relación entre el ser humano y los microorganismos que residen en él se ha multiplicado en los últimos años. Ya no denominamos a estos seres vivos «flora intestinal» ni los consideramos simplemente comensales. Los humanos somos, de hecho, «superorganismos» gobernados, en parte, por los microorganismos que hospedamos1. Los objetivos de esta revisión son acercarnos a los términos empleados actualmente en el boyante campo de la microbiota humana, en particular de la microbiota intestinal, conocer las profundas implicaciones de la dieta y el medio ambiente en la microbiota normal y anormal, y por último, esbozar un panorama de la relación entre la microbiota y las enfermedades gastrointestinales.
Se realizó una revisión de la literatura por medio de la consulta de las bases de datos de PubMed de los últimos 15 años, así como de los estudios presentados en la Digestive Diseases Week en San Diego, California, en 2012, y en la United European Gastroenterology Week en Amsterdam, en el mismo año.
Microbiota y otros conceptosActualmente se utilizan una serie de términos con los cuales conviene que nos familiaricemos. El término microbiota hace referencia a la comunidad de microorganismos vivos residentes en un nicho ecológico determinado. La microbiota residente en el intestino humano es una de las comunidades más densamente pobladas2, incluso más que el suelo, el subsuelo y los océanos. En el intestino grueso de los mamíferos la cifra de microorganismos se eleva a 1012-1014. Este número es mayor, incluso, que el de células humanas3. El ecosistema microbiano del intestino (microbiota intestinal) incluye muchas especies nativas que colonizan permanentemente el tracto gastrointestinal, y una serie variable de microorganismos que solo lo hacen de manera transitoria. Al conjunto formado por los microorganismos, sus genes y sus metabolitos se le denomina microbioma. El microbioma humano se refiere a la población total de microorganismos con sus genes y metabolitos que colonizan el cuerpo humano, incluyendo el tracto gastrointestinal, el genitourinario, la cavidad oral, la nasofaringe, el tracto respiratorio y la piel4. El Proyecto del Microbioma Humano ha identificado aproximadamente al 30% de la microbiota intestinal5, y junto con el proyecto Metagenómica del Tracto Intestinal Humano en Europa, y muchos otros grupos, trabaja activamente para identificar a todos los genes de la microbiota.
A las alteraciones de la microbiota intestinal y la respuesta adversa del hospedero a estos cambios se le ha denominado disbiosis. La disbiosis se ha asociado con afecciones tan disímiles como el asma, las enfermedades inflamatorias crónicas, la obesidad y la esteatohepatitis no alcohólica (EHNA)6–8.
El estudio del microbioma ha presentado varios retos en el pasado: no todos los microorganismos son fáciles de cultivar. Sin embargo, las técnicas modernas de estudio del material genético han revolucionado nuestro conocimiento del microbioma. Algunos componentes de la microbiota requieren condiciones especiales para su cultivo, por lo que tradicionalmente no eran detectados o no se conocían. Por ejemplo, la microbiota colónica tiene aproximadamente de 800 a 1.000 especies por individuo, pero el 62% de ellas no se conocían y el 80% de las bacterias identificadas por la metagenómica se consideran no cultivables9.
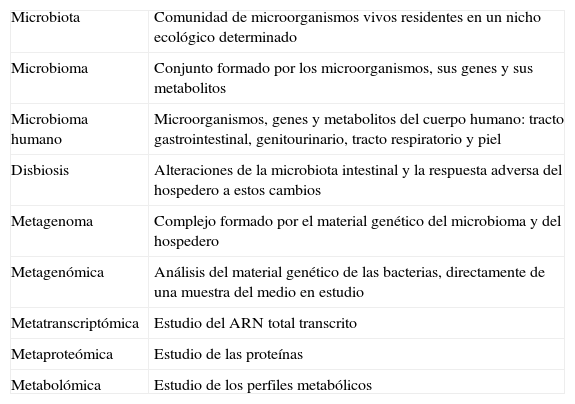
Los conceptos y avances en la «meta-ómica» aportan una ventana para comprender a la microbiota intestinal10 (tabla 1):
- -
La metagenómica es el análisis del material genético de las bacterias, directamente de una muestra del medio en estudio, con lo que se puede identificar a las bacterias que no se detectan con los cultivos.
- -
La metatranscriptómica estudia al ARN total transcrito.
- -
La metaproteómica se enfoca en los niveles de proteínas.
- -
La metabolómica estudia los perfiles metabólicos.
- -
El metagenoma es el complejo formado por los genes del hospedero y el microbioma.
Conceptos sobre la microbiota
| Microbiota | Comunidad de microorganismos vivos residentes en un nicho ecológico determinado |
| Microbioma | Conjunto formado por los microorganismos, sus genes y sus metabolitos |
| Microbioma humano | Microorganismos, genes y metabolitos del cuerpo humano: tracto gastrointestinal, genitourinario, tracto respiratorio y piel |
| Disbiosis | Alteraciones de la microbiota intestinal y la respuesta adversa del hospedero a estos cambios |
| Metagenoma | Complejo formado por el material genético del microbioma y del hospedero |
| Metagenómica | Análisis del material genético de las bacterias, directamente de una muestra del medio en estudio |
| Metatranscriptómica | Estudio del ARN total transcrito |
| Metaproteómica | Estudio de las proteínas |
| Metabolómica | Estudio de los perfiles metabólicos |
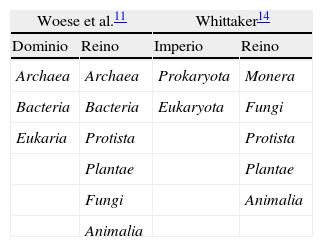
Se han descrito varios sistemas de clasificación de los reinos biológicos (tabla 2). En 1990 Woese et al. introdujeron el término «dominio» para sustituir al de «reino» como orden taxonómico superior, dividiendo a todos los seres vivos en Bacteria, Archaea y Eukarya11. Las arqueas, organismos unicelulares, antes agrupadas en el reino de las bacterias, tienen un material genético lo suficientemente distinto al de las bacterias como para clasificarlas en un dominio separado12. Un descubrimiento reciente es la presencia en la microbiota intestinal de miembros del dominio Archaea, considerado actualmente distinto al dominio Bacteria. Un ejemplo de arqueas es el Methanobrevibacter smithii, productor de metano (CH4), implicado en estudios recientes en el síndrome de intestino irritable (SII) con estreñimiento13.
El ARN ribosomal (ARNr) es la macromolécula más ampliamente usada en estudios de filogenia y taxonomía bacteriana15. La secuenciación de las regiones variables del gen que codifica para la subunidad 16S del ARNr (ARNr 16S) identifica el parecido filogenético de las bacterias y las arqueas, y permite clasificarlas sin el uso de cultivos. La información genética obtenida del microbioma por medio del ARNr 16S se agrupa en las llamadas unidades taxonómicas operacionales, de acuerdo con el porcentaje de semejanza de sus ARNr 16S. Cuando hay una semejanza en el ARNr 16S del 95% se habla de género, y cuando la semejanza es del 97%, se habla de especie16.
Alrededor del 50% de la masa fecal está constituida por bacterias. Esta población se compone de trillones de microorganismos pertenecientes, fundamentalmente, a 4 filas: Firmicutes, Bacteroidetes, Actinobacteria y Proteobacteria, con un predomino de las 2 primeras (90%)17.
Funciones de la microbiotaLa microbiota intestinal ha pasado de considerarse un comensal acompañante, a considerarse un «órgano metabólico»18, con funciones en la nutrición, la regulación de la inmunidad y la inflamación sistémica19. Los mamíferos que crecen libres de gérmenes (LG) tienen un desarrollo corporal anormal, con pared intestinal atrófica, corazón, pulmones e hígado de bajo peso y sistema inmune inmaduro con niveles bajos de inmunoglobulinas20. Backhed et al. demostraron que un grupo de ratones tenía un 40% más de grasa corporal que sus contrapartes LG sometidos a la misma dieta21, y que los ratones LG están protegidos de la obesidad ocasionada por dietas altas en grasa y azúcar22. Cuando a los ratones LG se les trasplanta microbiota obtenida del ciego de ratones normales («convencionalizar») ocurre un incremento significativo de su contenido de grasa corporal21. La microbiota intestinal tiene enzimas que transforman a los polisacáridos complejos de la dieta, que el intestino humano no puede digerir ni absorber, en monosacáridos y ácidos grasos de cadena corta (AGCC), principalmente acético, propiónico y butírico. Los 2 primeros se absorben a la circulación portal y el tercero es empleado por los colonocitos como fuente de energía. Los AGCC pueden ser transportados al hígado para ser usados en la síntesis lipídica; de hecho, se estima que las calorías derivadas de esta digestión bacteriana constituyen alrededor del 10% de toda la energía que absorbemos23. La cantidad de AGCC en el colon y en la sangre son importantes para la inmunorregulación del hospedero. Algunos estudios reportan efectos positivos de los AGCC en pacientes con alteraciones inflamatorias del intestino, de hecho, dichos individuos tienen concentraciones mucho menores de AGCC24–26. Además, parece ser que la microbiota es capaz de modular los genes que afectan la disposición de la energía en los adipocitos2. Los microbios y los vertebrados evolucionaron juntos a través de miles de años, y el funcionamiento normal del sistema digestivo e inmunológico depende de la presencia de la microbiota simbiótica27.
Factores que influyen sobre la microbiotaEvolutivamente, en los mamíferos, los organismos que componen la microbiota son determinados por los tipos de fuentes nutricionales, siendo diferentes los perfiles de omnívoros, carnívoros y herbívoros28. Las características de la dieta, junto con los factores genéticos, influyen en el predominio de unos microorganismos sobre otros29. Después de tan solo un día de dieta de tipo occidental (alta en grasa y azúcar y baja en polisacáridos de las plantas), los ratones muestran cambios en su composición microbiana y en sus vías metabólicas, y en 2 semanas han desarrollado más adiposidad30. La abundancia o escasez de alimento determinará la presencia o no de especies bacterianas que se reproducen bien cuando hay disponibilidad ilimitada de alimentos, o de especies más eficientes cuando los nutrientes son escasos29,31. Los ratones sometidos a dietas de tipo occidental muestran un incremento de Firmicutes y una disminución de Bacteroidetes30.
In utero, el ser humano carece de microbiota. Al nacer, el tracto gastrointestinal se coloniza inmediatamente. Hasta la vía de nacimiento (parto o cesárea) y el tipo de alimentación (seno materno o fórmula) ha demostrado producir diferencias en la microbiota intestinal32. Los perfiles fecales microbianos del lactante muestran un parecido marcado con los perfiles bacterianos del canal de parto y de la leche materna33. Durante la infancia y a lo largo de la vida, la composición microbiana también cambia de acuerdo con la edad y la dieta34. En los primeros 2 años de vida, la microbiota está dominada por las bifidobacterias35. Posteriormente, la composición microbiana se diversifica y alcanza su máxima complejidad en el adulto, con cientos de filotipos dominados por Bacteroidetes y Firmicutes36.
Aun cuando la microbiota intestinal cambia con el paso de los años, el medio ambiente y la microbiota materna durante el parto y la alimentación al seno parecen permanecer como factores muy importantes en el desarrollo de la microbiota en el futuro. Una vez establecida la microbiota en un individuo, es estable en el tiempo37. En humanos, los miembros de la misma familia tuvieron comunidades bacterianas más parecidas entre ellos que en comparación con individuos no relacionados38.
Arumugam et al. enunciaron recientemente el concepto de los enterotipos, con la idea de clasificar las distintas microbiotas de intestino humano, con base en la composición de sus comunidades bacterianas y de acuerdo con la abundancia de los diversos géneros bacterianos39. Esto podrá facilitar la asociación de los distintos enterotipos con las diversas condiciones asociadas a la disbiosis. Estudios como este nos ayudarán a desenmarañar los datos con los que contamos, y poder correlacionar las distintas poblaciones microbianas con las entidades clínicas producto de la disbiosis.
Sin embargo, debemos tener cuidado al sacar conclusiones, y pensar que los cambios en la microbiota siempre son la causa y no la consecuencia de las modificaciones en la fisiología de los organismos. Por ejemplo, Purna et al. demostraron que los efectos en la microbiota asociados a la introducción de fibra en la dieta podrían estar relacionados con la velocidad del tránsito intestinal y no con la fibra misma, pues reprodujeron los mismos cambios en la microbiota al utilizar laxantes inertes40.
Microbiota e inmunidadLa microbiota intestinal ejerce un importante efecto sobre la respuesta inmune del humano. En 1989, Strachan mostró que la disminución en la carga microbiana debida a la elevación de los estándares de higiene en los países desarrollados podría conducir a un incremento de las enfermedades autoinmunes41. La dieta y los efectos de esta en la microbiota intestinal y en la respuesta inmune se han postulado como posibles explicaciones para el incremento en la incidencia de enfermedades inflamatorias como el asma y la diabetes tipo 1 en los países desarrollados27. Nuevos hallazgos sobre la microbiota intestinal y su capacidad inmunomoduladora coinciden con los datos epidemiológicos que conectan la obesidad y el asma o la obesidad y la diabetes tipo 142,43.
La mucosa intestinal ejerce funciones de inmunidad adaptativa ya que su sistema inmune tiene la capacidad de responder a una infinidad de antígenos, pero también existe la inmunidad innata que es el reconocimiento de determinados antígenos, y que es heredada filogenéticamente desde las plantas hasta los vertebrados. Estos antígenos se han llamado patrones moleculares asociados a patógenos (PMAP) e incluyen lípidos, lipopolisacáridos (LPS) y lipoproteínas. Los PMAP son reconocidos por los receptores de reconocimiento de patrones. La interacción entre estos y los PMAP induce la producción de citocinas e interferones. Entre otros, los receptores de reconocimiento de patrones incluyen a los receptores tipo Toll (TLR), que son receptores transmembrana. Varios de los PMAP que son ligandos de los TLR contienen lípidos, indispensables para su actividad agonista, como los LPS bacterianos (endotoxinas bacterianas), ligandos de los TLR-4. Los LPS son componentes esenciales de la pared celular bacteriana. Aunque no son estrictamente factores de virulencia bacterianos, despiertan una intensa respuesta de la inmunidad innata. Los TLR se expresan en las células encargadas de la inmunidad innata, como macrófagos, células epiteliales, células endoteliales y adipocitos, y en el parénquima de algunos órganos44, pero también en células de la inmunidad adaptativa, que incluyen a las células B, los mastocitos, las células T y las células dendríticas, las cuales son clave para el inicio de la inmunidad adaptativa44. Las células dendríticas son un tipo de células presentadoras de antígenos, están ubicadas en la lámina propia, extienden sus apéndices entre las células epiteliales de la mucosa y muestran patrones moleculares de microorganismos patógenos y comensales45. Las señales originadas en los TLR inducen a las células dendríticas a diferenciarse y a producir citocinas46. Las células dendríticas presentan los antígenos a las células T y están implicadas tanto en funciones de defensa como de tolerancia inmunológica a los alimentos y a los microorganismos47.
Cuando los LPS se unen a los TLR-4 se produce una respuesta inflamatoria intensa con daño al tejido blanco. Los LPS se detectan en la circulación de individuos sanos y sus niveles se incrementan después de la ingestión de alimentos ricos en energía48.
Hasta hace poco se consideraba al tejido adiposo como un mero compartimento de almacenaje, sin embargo, el adipocito es una célula endocrina activa productora de adipocinas49. En la obesidad, además del incremento del volumen del adipocito, el tejido adiposo está infiltrado de macrófagos. Estos tienen 2 subpoblaciones: M1, que producen citocinas inflamatorias, y M2, que generan productos antiinflamatorios50. Los TLR promueven el fenotipo M1, con el consiguiente incremento de citocinas proinflamatorias.
La «teoría de la higiene» supone que el exceso de limpieza y la disminución en la exposición a las bacterias a temprana edad impide el correcto desarrollo de los mecanismos inmunorreguladores, que previenen las respuestas inapropiadas de las células T y las enfermedades inflamatorias posteriores27. Hansen et al. demostraron que los ratones LG que se «convencionalizan» a las 3 semanas de edad con contenido cecal de ratones normales modifican permanentemente la composición de su microbiota intestinal y desarrollan una respuesta inmune proinflamatoria, es decir, el corto periodo posnatal libre de gérmenes ejerció cambios adversos y permanentes en la inmunidad51. De manera interesante, si la convencionalización se realizaba en la primera semana de vida, no se reproducían estos efectos, lo que hace pensar que hay una ventana de tiempo en la cual se puede modificar permanentemente la inmunidad.
Hay diferencias radicales entre la microbiota intestinal de niños en África y la de niños en zonas urbanas de Europa. Los niños de Burkina Faso (África) tienen una dieta muy alta en fibra y su microbiota tiene grandes cantidades de Bacteroidetes, que hidrolizan los polisacáridos complejos de las plantas, y tienen mucha menor abundancia de Firmicutes que la microbiota de una cohorte europea52. Es interesante saber que las alergias y el asma son prácticamente inexistentes en las comunidades rurales de África.
La evidencia que apunta hacia una alteración de la microbiota intestinal en personas con alergia y asma se acumula24. Los niños que viven en granjas tienen una incidencia menor de asma que los niños de la ciudad53.
Microbiota y metabolismoLa obesidad resulta del incremento en el consumo de alimentos altos en energía, azúcares y grasas saturadas, sin embargo, parece ser que el simple incremento en la ingestión de calorías no explica completamente la actual epidemia de obesidad. Los ratones LG no aumentan su peso cuando se exponen a dietas altas en grasa y en hidratos de carbono, lo que hace suponer que la dieta no es suficiente para inducir la obesidad.
Se ha descrito una microbiota humana de «tipo obeso», asociada al exceso de peso y al síndrome metabólico, con un incremento de la razón Firmicutes/Bacteroidetes54. Las Bifidobacteria y los Bacteroides spp. parecen ser protectores contra el desarrollo de obesidad55. Esta podría tener un componente microbiano, con probables implicaciones terapéuticas.
La colonización de ratones LG con la microbiota de ratones normales produce un incremento dramático de la grasa en 10-14 días, a pesar de una disminución en el consumo de alimentos. La capacidad para fermentar hidratos de carbono de la dieta varía ampliamente entre microorganismos y las evidencias apuntan hacia una mayor eficiencia de la microbiota intestinal de los individuos con sobrepeso para degradar los hidratos de carbono no digeribles de los vegetales23. Turnbaugh et al.23 demostraron que los ratones genéticamente obesos (ob/ob) tienen un 50% menos de Bacteroidetes y más Firmicutes que sus hermanos delgados. Comprobaron que la microbiota de los ratones obesos liberaba más calorías durante la digestión que la de los delgados. El fenotipo generador de obesidad puede ser transmisible: la implantación de la microbiota intestinal obesogénica en ratones LG trae como resultado una adiposidad incrementada en el ratón receptor23.
Cuando se suministra a ratones con peso normal una dieta típica occidental elevada en calorías durante 8 semanas (aceptado mecanismo de generación de obesidad en ratones) se observa también una marcada reducción de Bacteroidetes y una manifiesta elevación de Firmicutes56. Jumpertz et al. administraron a 12 personas delgadas y 9 obesas dietas variables en contenido calórico y compararon las calorías ingeridas con las calorías fecales. La modificación en la microbiota secundaria a la dieta, con un incremento del 20% de Firmicutes y la correspondiente disminución de Bacteroidetes, se asoció con un incremento en la recuperación de energía de aproximadamente 150kcal57.
Estos hallazgos han llevado a la hipótesis de que la microbiota de los individuos obesos puede ser más eficiente en la extracción de energía que la microbiota de los individuos delgados.
Se sabe que situaciones que ocurren alrededor del nacimiento incrementan el riesgo de desarrollar obesidad, diabetes y enfermedad cardiovascular en la etapa adulta58, y la colonización inicial podría ser muy importante para determinar la composición final de la microbiota permanente en los adultos59.
Algunos de los múltiples mecanismos metabólicos que asocian la microbiota con la obesidad y con sus trastornos relacionados, como la diabetes y el hígado graso, son los siguientes:
- -
La fermentación bacteriana de los polisacáridos de la dieta, que no pueden ser digeridos por el hospedero, con la consecuente producción de monosacáridos y AGCC. Los AGCC son sustratos de los colonocitos y precursores del colesterol y los ácidos grasos, son sustratos de la gluconeogénesis en el hígado, todo lo cual optimiza el aprovechamiento de la energía de la dieta.
- -
Los AGCC se unen a receptores específicos de células intestinales endocrinas (GRP43 y GRP41) que incrementan el péptido YY, el cual retarda el tránsito intestinal, aumentando la absorción de nutrientes60, e incrementan los niveles de leptina, una hormona orexigénica61.
- -
La regulación microbiana de algunos genes del hospedero que promueven el depósito de lípidos en los adipocitos21.
- -
La disminución de la expresión intestinal del factor adiposo inducido por el ayuno (FIAF), también conocido como factor tipo iv parecido a la angiopoyetina, que es un inhibidor circulante de la lipasa lipoproteica, que favorece la captura de los ácidos grasos y la expansión del tejido adiposo. El FIAF también induce al coactivador 1 del receptor activado gamma de proliferación de peroxisomas, que regula la expresión de las enzimas encargadas de la oxidación de los ácidos grasos22. De hecho, los ratones LG que carecen de los 2 alelos de FIAF tienen la misma cantidad de grasa corporal que los ratones convencionales21, por lo que se piensa que FIAF puede ser un mediador de la regulación microbiana de las reservas periféricas de grasa62.
- -
Los ratones obesos tienen un incremento de las arqueas metanogénicas, lo que se asocia con una presión parcial de hidrógeno menor, que optimiza la velocidad de fermentación bacteriana63,64.
- -
El incremento hepático de la captura de monosacáridos de la circulación portal activa factores transcripcionales clave como ChREBP, que regulan la lipogénesis65.
- -
La microbiota incrementa la vascularización inducida por inflamación y el flujo sanguíneo de la mucosa, lo que aumenta la absorción de nutrientes66.
- -
La microbiota intestinal es capaz de promover un estado de inflamación sistémica de bajo grado, resistencia a la insulina e incrementar el riesgo cardiovascular a través de mecanismos que incluyen la exposición a productos bacterianos, en particular, los LPS derivados de las bacterias gramnegativas. A esto se le ha llamado endotoxemia metabólica67. Clemente-Postigo et al. demostraron recientemente una asociación entre los niveles de triglicéridos posprandiales y la elevación de endotoxinas bacterianas después de una dieta alta en grasa68. Los cambios en la microbiota intestinal, el aumento de la permeabilidad intestinal y la endotoxemia posiblemente juegan un papel en el desarrollo de un estado inflamatorio crónico de bajo grado en el hospedero que contribuye al desarrollo de la obesidad y de enfermedades metabólicas crónicas, como el hígado graso no alcohólico (HGNA)67,69.
- -
En años recientes, se ha restado importancia al IMC como predictor del síndrome metabólico y ha ganado fuerza el concepto de que la grasa visceral es la responsable de este problema. La grasa visceral secreta cerca de 250 proteínas2, como el factor de crecimiento visceral, la IL-6, el inhibidor del activador de plasminógeno, el TNF-α y la proteína C reactiva, todos implicados en la inflamación70. Esto hace pensar que la obesidad, con sus consecuencias metabólicas y enfermedades acompañantes, podría tener un importante componente microbiano, con probables implicaciones terapéuticas.
Estudios recientes empiezan a perfilar la asociación entre la disbiosis y las enfermedades gastrointestinales. Se han demostrado diferencias importantes en la microbiota de los pacientes con SII en comparación con los controles sanos; en los pacientes se demostró un incremento de 2 veces en la relación Firmicutes/Bacteroidetes (P<.0002)71. Los pacientes con SII tienen menos Lactobacillus y Bifidobacterium spp. que los controles sanos72. Las bacterias antes mencionadas se unen a las células epiteliales e inhiben la adherencia de bacterias patógenas, no producen gas al fermentar los hidratos de carbono e inhiben a los Clostridia spp.73. Los probióticos modifican la fermentación colónica y estabilizan la microbiota colónica. Varios estudios con probióticos han demostrado una mejoría en la flatulencia y la distensión abdominal74. Hay interesantes hallazgos en estudios recientes sobre SII en adultos y en niños. Saulnier et al. encontraron un porcentaje significativamente mayor de proteobacterias en niños con SII. Pudieron clasificar los subtipos de SII con base en una serie limitada de bacterias. De manera interesante, un nuevo microbio parecido a Ruminococcus se asoció con el SII75.
Estudios realizados en la década pasada identificaron una asociación entre el SII y la sobrepoblación bacteriana detectada por medio de pruebas de aliento con administración de lactulosa o glucosa por vía oral. Pimentel et al. demostraron, en pacientes con SII, un 35% de mejoría de los síntomas al administrar un antibiótico no absorbible (neomicina), en comparación con un 11.4% de mejoría al administrar placebo. Cuando tomaron en cuenta solamente a los pacientes en quienes se demostró la eliminación de la sobrepoblación bacteriana después del uso del antibiótico, la mejoría ocurrió en el 75% de ellos76. Esta línea de investigación ha sido tomada con cautela, debido a las dificultades para diagnosticar la sobrepoblación bacteriana por medio de pruebas de aliento. Además, el uso de antibióticos con poca absorción, como la neomicina, no está exento de efectos secundarios. Más recientemente, se reportaron 2 estudios fase 3, doble ciego y controlados con placebo (TARGET 1 y TARGET 2) en pacientes con SII sin estreñimiento tratados con rifaximina, un antibiótico no absorbible, a dosis de 550mg 3 veces al día por 2 semanas, para investigar la mejoría de los síntomas del SII. Significativamente más pacientes en el grupo de rifaximina tuvieron una mejoría global de los síntomas de SII, 40.7 contra 31.7% (P=.01), y mejoría en la sensación de distensión abdominal, 40.2 contra 30.3% (P=.001)77.
Son sorprendentes los estudios que demuestran que existe un eje intestino-cerebro-microbiota78. Se ha demostrado que el contenido microbiano del tracto gastrointestinal posnatal en ratones es crítico para el desarrollo de respuestas adecuadas al estrés en etapas posteriores de la vida. También se ha demostrado que existe una ventana crítica en las etapas tempranas de la vida en la que debe ocurrir la colonización para asegurar un desarrollo normal del eje hipotálamo-hipófisis-adrenal79.
El CH4 es uno de los gases presentes en el intestino humano y es producido por la fermentación bacteriana anaerobia. Se ha descrito que el CH4 puede afectar la velocidad del tránsito intestinal, reducir la secreción de serotonina y se ha asociado con el SII, la diverticulosis y el cáncer de colon80. El principal microorganismo productor de CH4 es Methanobrevibacter smithii, perteneciente al dominio Archaea81. Se han demostrado tiempos de tránsito intestinal prolongados en adultos productores de CH482. Un estudio reciente evaluó la producción de CH4 en 629 pacientes con síntomas intestinales por medio de una prueba de aliento con glucosa. El 32.3% de los pacientes eran productores de CH4. La excreción de este gas se pudo correlacionar significativamente con el estreñimiento crónico y fue más alta en pacientes con estreñimiento en comparación con los individuos sanos, y mucho mayor que la de los pacientes con diarrea83.
Enfermedad de CrohnMuchos estudios han mostrado la presencia de disbiosis en el intestino de pacientes con enfermedad de Crohn en comparación con los individuos sanos84. Los gemelos sanos suelen tener una microbiota intestinal muy parecida, pero cuando uno de los gemelos tiene enfermedad de Crohn, la composición intestinal cambia mucho, sobre todo en pacientes con inflamación ileal85.
Enfermedad celíacaUn marcador de la enfermedad celíaca activa es la producción de citocinas por los linfocitos T intestinales en individuos acarreadores de ciertos alelos del MHC clase ii. Se ha propuesto que la disbiosis es otro factor de riesgo para la enfermedad celíaca. De hecho, se describió una «epidemia sueca de enfermedad celíaca»86, y se han aislado bacterias candidatas como factores etiológicos, que posteriormente se han podido aislar en pacientes nacidos durante la epidemia. La disbiosis y las bacterias asociadas a la enfermedad celíaca pueden ser un factor de riesgo para el desarrollo del padecimiento, ya sea por influencia directa en las respuestas inmunes de la mucosa o al incrementar la respuesta inflamatoria al gluten87.
Esteatohepatitis no alcohólica/hígado graso no alcohólicoAl convencionalizar ratones LG a partir del ciego de ratones normales, Backhed et al. demostraron que aumenta la grasa hepática21. La EHNA y el HGNA se han asociado con la sobrepoblación bacteriana y el incremento de la permeabilidad intestinal, aunque no todos los estudios son concordantes62.
Varios productos bacterianos pueden ser potencialmente hepatotóxicos: fenoles, amoniaco, etanol y otros88. Se ha descrito un incremento en la producción de etanol en pacientes obesos89. Se piensa que el principal producto bacteriano implicado en la EHNA y el HGNA es el LPS, componente activo de las endotoxinas de la pared bacteriana, liberado con la muerte bacteriana en el intestino. El LPS sufre traslocación capilar por medio de un mecanismo dependiente de TLR-4 y es absorbido junto con los lípidos de la dieta90. La absorción del LPS activa a su vez la producción de TNF-α, IL-1 e IL-6. Las señales que despierta el TLR-4 promueven la resistencia a la insulina, la esteatosis hepática, la inflamación y la fibrogénesis88.
La infusión crónica de dosis bajas de LPS en ratones provoca obesidad y un incremento en el porcentaje de grasa corporal, resistencia a la insulina, infiltración de macrófagos en el tejido adiposo y esteatosis hepática91. Estudios en humanos también han demostrado que la endotoxemia es un factor de riesgo para el desarrollo de EHNA/HGNA. Dos estudios con pacientes con HGNA diagnosticado con biopsia demostraron aumento de la endotoxemia en comparación con individuos sanos92,93.
En conclusión, el análisis moderno del genoma bacteriano es sumamente interesante, y ha abierto un campo de investigación que puede explicarnos la relación cercana entre el microbioma y el ser humano y puede ayudar a resolver cuestionamientos sobre las «epidemias» modernas: enfermedades autoinmunes, alérgicas y metabólicas, pero, sobre todo, nos ofrece la posibilidad de intentar revertirlas por medio de la manipulación de los componentes de la microbiota. La evidencia demuestra que la microbiota es estable durante el tiempo, y que algunos efectos de la colonización del ser humano en etapas tempranas no son reversibles. Algunas preguntas: ¿tendremos la capacidad de evitar las alteraciones en la microbiota debidas al exceso de higiene y la falta de contacto con los microorganismos saludables?, ¿podemos manipular la microbiota de un individuo de manera permanente o por lo menos a largo plazo?
FinanciaciónNo hubo financiamiento para la realización de este artículo.
Conflicto de interesesLa autora no tiene conflicto de interés en relación con el artículo que se remite para publicación.